Code
source("script.R")Determinantes de la esperanza de vida femenina
En este caso, actuamos como analistas de datos para una organización internacional. Nuestro objetivo es explicar qué factores estructurales (económicos y sanitarios) determinan la esperanza de vida de las mujeres (lifeexpf) en 122 países.
Las variables predictoras disponibles son:
urban: % Población urbana.docs: Médicos por 10.000 hab.hospbed: Camas hospitalarias por 10.000 hab.gdp: PIB per cápita ($).radio: Radios por 100 hab. (Indicador de acceso a información/tecnología).source("script.R")Retenemos los 116 países de los cuáles tenemos información completa.
La regresión lineal exige, como su nombre indica, que la relación entre la variable dependiente (\(Y\)) y las independientes (\(X\)) sea una línea recta, es por ello que aunque no requisito esencial, siempre es bueno analizar la relación de cada independiente con la dependiente y ver si se requiere linealizar para mejorar el que el modelo sea lineal.
# Matriz de dispersión
pairs(select(data, lifeexpf, urban, docs, hospbed, gdp, radio),
main = "Matriz de dispersión (Datos originales)",
pch=19, col="steelblue", cex=0.1)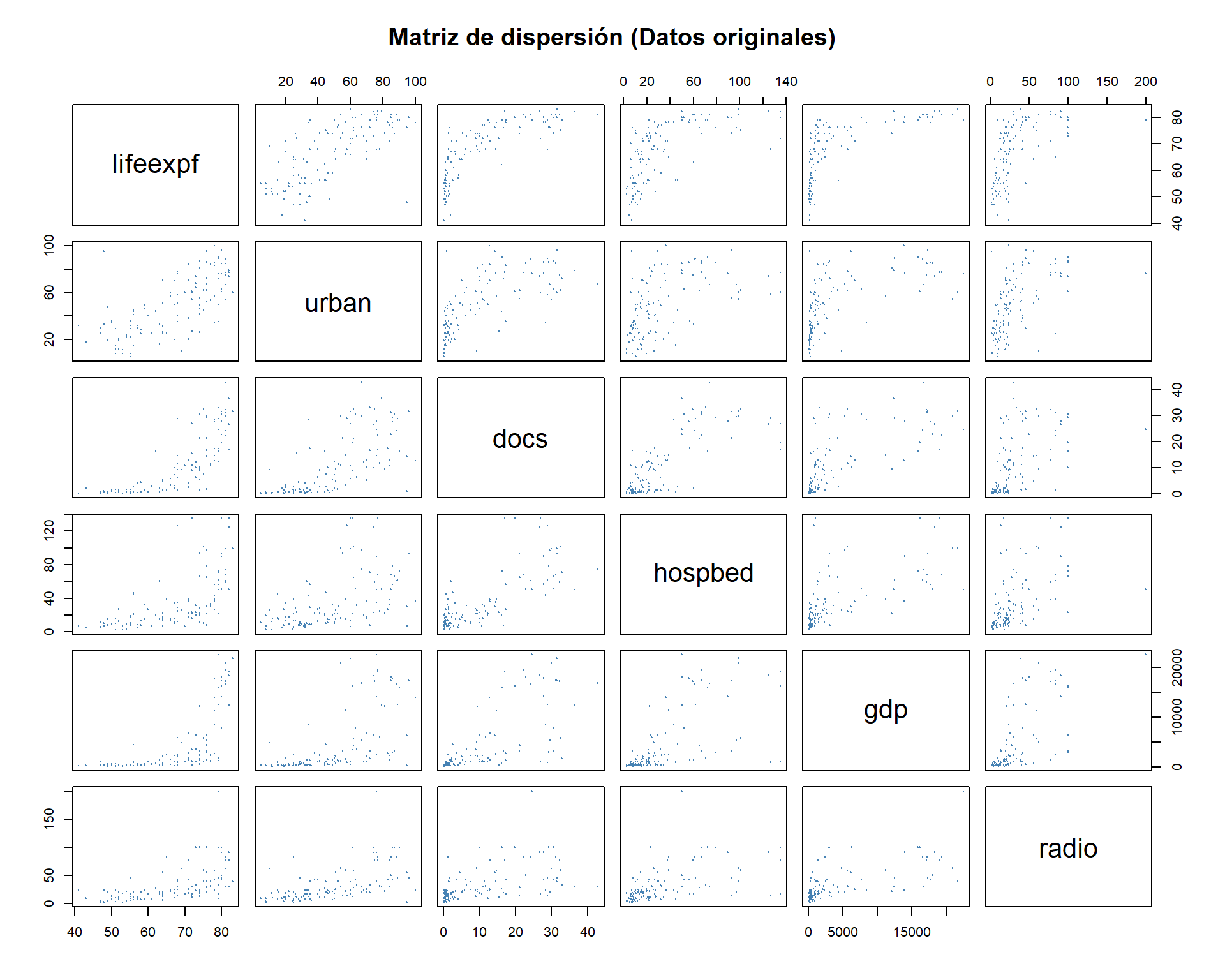
Diagnóstico: Observamos claramente que las relaciones no son lineales. * Fíjate en el cruce de lifeexpf (eje Y) con gdp o docs (eje X): forman curvas logarítmicas. * La lógica económica: El impacto de los primeros dólares de PIB en la vida es enorme (comida, agua), pero a partir de cierto nivel, ganar más dinero apenas alarga la vida (rendimientos decrecientes). * Ajustar una recta aquí sería un error grave: subestimaríamos la esperanza de vida en países pobres y ricos, y la sobreestimaríamos en los medios.
Para corregir esto, aplicamos logaritmos neperianos a las variables independientes. Esto “estira” los valores bajos y “comprime” los altos, enderezando la curva.
# Transformación Logarítmica
# Sumamos una pequeña constante (+1) por seguridad si hubiera ceros
data_model <- data %>%
mutate(
ln_urban = log(urban + 1),
ln_docs = log(docs + 1),
ln_hospbed = log(hospbed + 1),
ln_gdp = log(gdp + 1),
ln_radio = log(radio + 1)
)
# Verificación visual
pairs(select(data_model, lifeexpf, ln_urban, ln_docs, ln_hospbed, ln_gdp, ln_radio),
main = "Matriz de dispersión (Variables transformadas)",
pch=19, col="darkgreen", cex=0.1)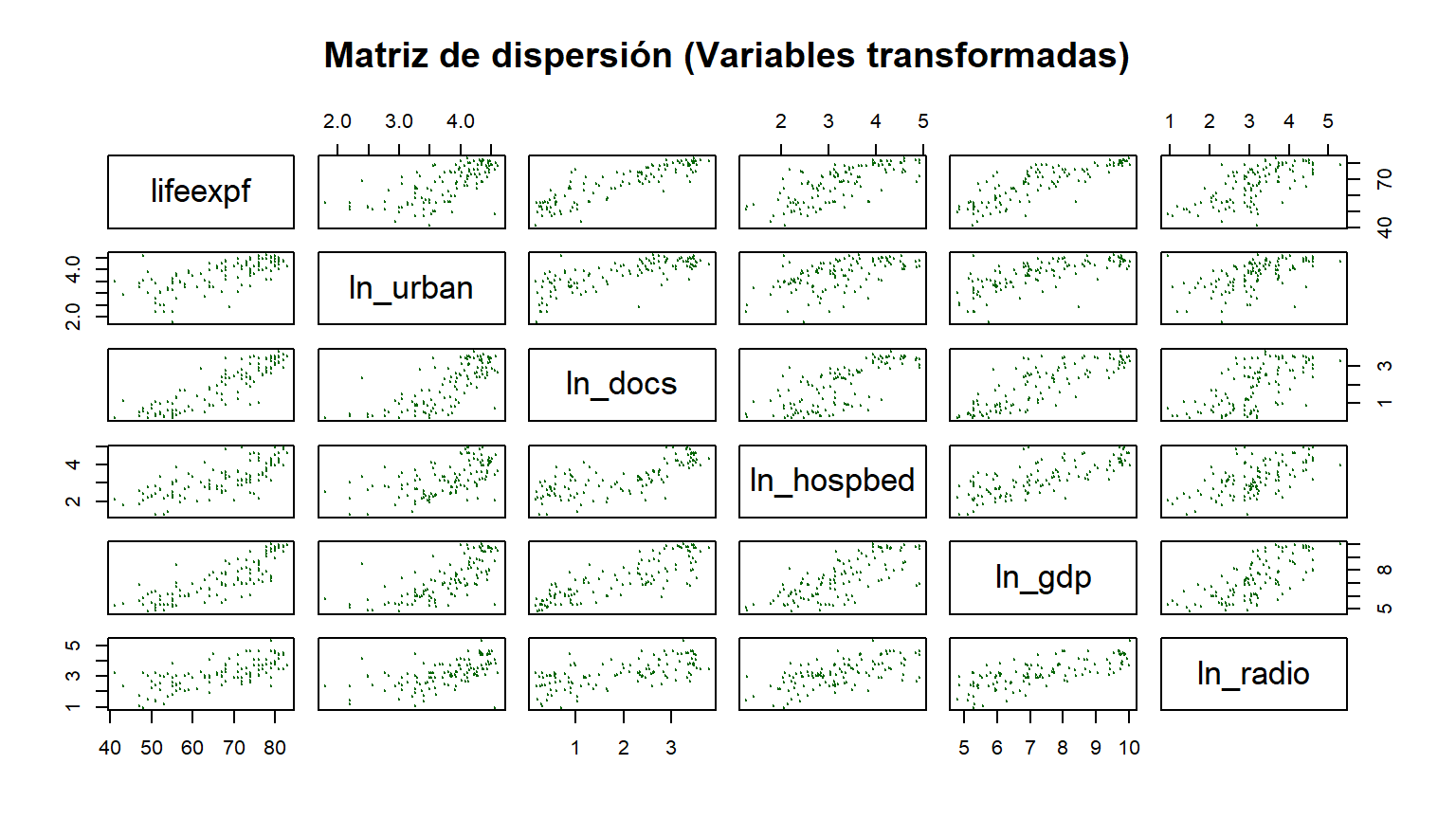
Conclusión: Ahora las nubes de puntos se asemejan mucho más a elipses o líneas rectas. La transformación ha sido exitosa y podemos proceder con el modelo.
Debemos asegurarnos de que no hay países con comportamientos tan extremos que distorsionen nuestra visión. En regresión múltiple, un país puede no ser “raro” por su PIB o por sus médicos por separado, pero sí por la combinación de ambos.
Para detectar estos casos extraños usamos la Distancia de Mahalanobis.
# Seleccionamos variables numéricas independientes para el cálculo
vars_indep <- select(data_model, ln_urban, ln_docs, ln_hospbed, ln_gdp, ln_radio)
# Cálculo de la distancia de Mahalanobis
# (Mide cuán lejos está cada país del "centro" multidimensional de los datos)
mh <- mahalanobis(vars_indep, colMeans(vars_indep, na.rm=TRUE), cov(vars_indep))
# Cálculo de la probabilidad (p-valor) asociada a esa distancia
# df = 5 (número de variables independientes)
pmh <- pchisq(mh, df=5, lower.tail=FALSE)
# Identificamos los casos extremos (Criterio estricto: p < 0.001)
outliers <- data[pmh < 0.001, ]
# Reporte de resultados
if (nrow(outliers)>0) {
print("Países detectados como outliers extremos por Mahalanobis:",outliers$country)
print("Estos casos se excluirían para la calibración del modelo.")
} else {
print("Países detectados como outliers extremos por Mahalanobis: Ninguno")
}[1] "Países detectados como outliers extremos por Mahalanobis: Ninguno"Debemos esperar a disponer del modelo para poder calcular la distancia de de Cook.
# Ajustamos el modelo con las variables transformadas
modelo1 <- lm(lifeexpf ~ ln_urban + ln_docs + ln_hospbed + ln_gdp + ln_radio, data = data_model)
distancia_cook <- cooks.distance(modelo1)
df_cook <- data.frame(
Country = data_model$country,
Distancia_Cook = distancia_cook) %>%
arrange(desc(Distancia_Cook))
print(paste0("Máxima Distancia de De Cook: ", max(df_cook$Distancia_Cook)))[1] "Máxima Distancia de De Cook: 0.0763275302552821"Como la máxima distancia de De Cook está por debajo de 1 , los ouliers detectados no influyen en el resultado del modelo. No se eliminan por tanto.
Veamos el modelo.
summary(modelo1)
Call:
lm(formula = lifeexpf ~ ln_urban + ln_docs + ln_hospbed + ln_gdp +
ln_radio, data = data_model)
Residuals:
female life expectancy
Min 1Q Median 3Q Max
-13.8126 -2.7923 -0.2122 2.5576 15.3557
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 36.8346 4.0647 9.062 5.49e-15 ***
ln_urban -0.3167 1.1330 -0.280 0.78033
ln_docs 5.3901 0.8220 6.557 1.84e-09 ***
ln_hospbed 0.9307 0.8318 1.119 0.26564
ln_gdp 1.7444 0.6248 2.792 0.00618 **
ln_radio 1.6334 0.7665 2.131 0.03531 *
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 4.874 on 110 degrees of freedom
Multiple R-squared: 0.8175, Adjusted R-squared: 0.8092
F-statistic: 98.52 on 5 and 110 DF, p-value: < 2.2e-16Interpretación del ajuste global: El modelo es excelente. Explica el 80% de la variabilidad en la esperanza de vida de las mujeres. * Prueba F (p < 2.2e-16): El modelo es estadísticamente significativo en su conjunto.
Obtenemos los parámetros estandarizados para evalluar su importancia.
lm.beta(modelo1)
Call:
lm(formula = lifeexpf ~ ln_urban + ln_docs + ln_hospbed + ln_gdp +
ln_radio, data = data_model)
Standardized Coefficients::
(Intercept) ln_urban ln_docs ln_hospbed ln_gdp ln_radio
NA -0.01762847 0.55401297 0.07303904 0.24091524 0.12813795 Para confiar en estos resultados, debemos auditar los residuos del modelo.
Comprobamos que la relación global sea lineal. Buscamos una nube aleatoria sin formas de “U”.
# Preparamos datos para ggplot
df_diag <- data.frame(
predichos = fitted(modelo1),
residuos_std = rstandard(modelo1)
)
ggplot(df_diag, aes(x = predichos, y = residuos_std)) +
geom_point(color = "steelblue", alpha = 0.6) +
geom_hline(yintercept = 0, linetype = "dashed") +
geom_smooth(method = "loess", se = FALSE, color = "red", linetype="dotted") +
labs(title = "Linealidad: Predichos vs. Residuos",
subtitle = "Buscamos una nube sin patrones curvos",
x = "Valores Predichos", y = "Residuos Estandarizados") +
theme_minimal()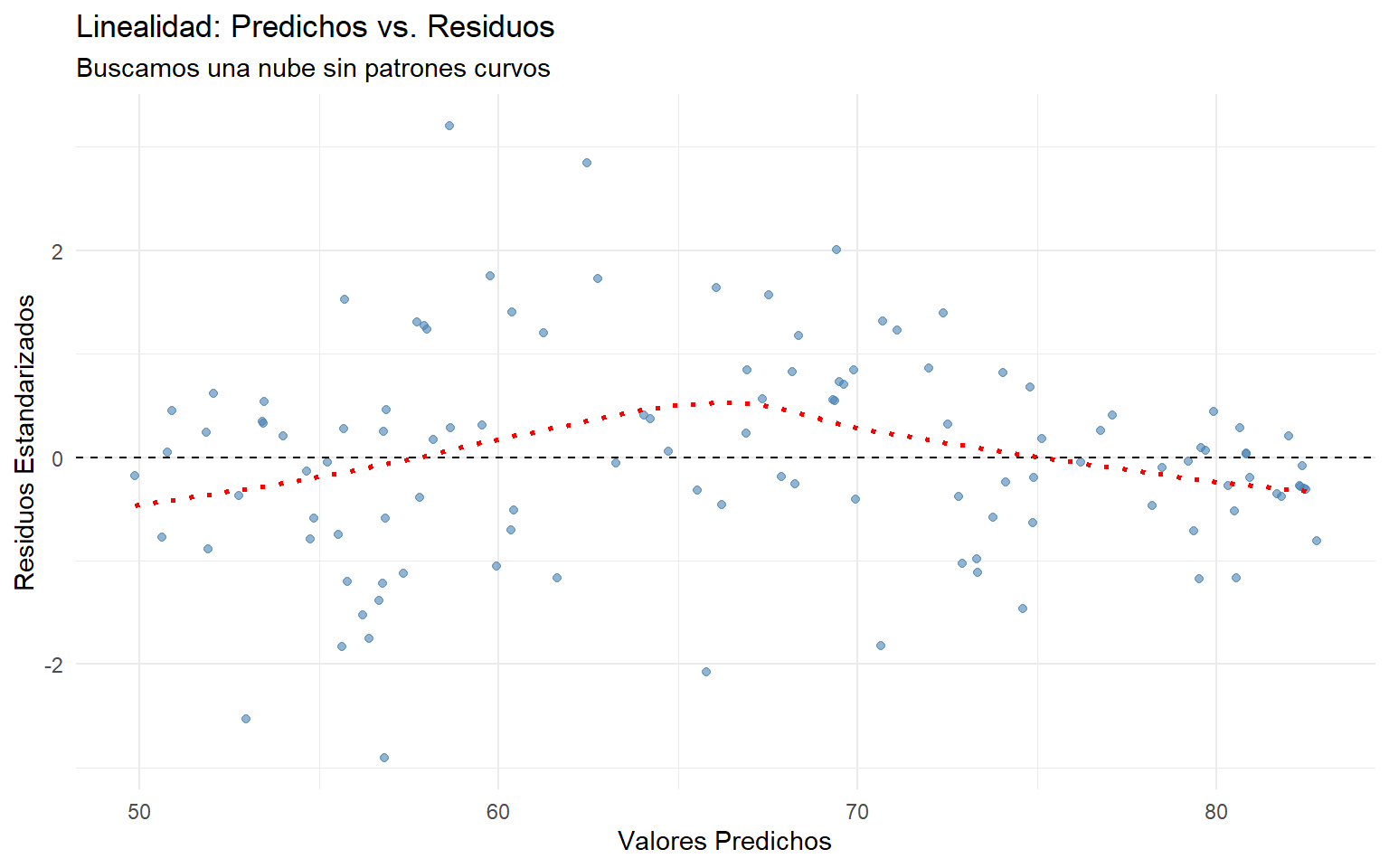
Los errores deben distribuirse siguiendo una campana de Gauss.
residuos <- residuals(modelo1)
p1 <- ggplot(data.frame(res = residuos), aes(x = res)) +
geom_histogram(aes(y = ..density..), bins = 15, fill = "steelblue", color = "white", alpha = 0.7) +
geom_density(color = "darkblue", size = 1) +
labs(title = "Histograma", subtitle = "¿Campana de Gauss?", x = "Residuos") + theme_minimal()
p2 <- ggplot(data.frame(res = residuos), aes(sample = res)) +
stat_qq(color = "steelblue") + stat_qq_line() +
labs(title = "Q-Q Plot", subtitle = "¿Hormigas en el alambre?", x = "Teórico", y = "Muestra") + theme_minimal()
p1 + p2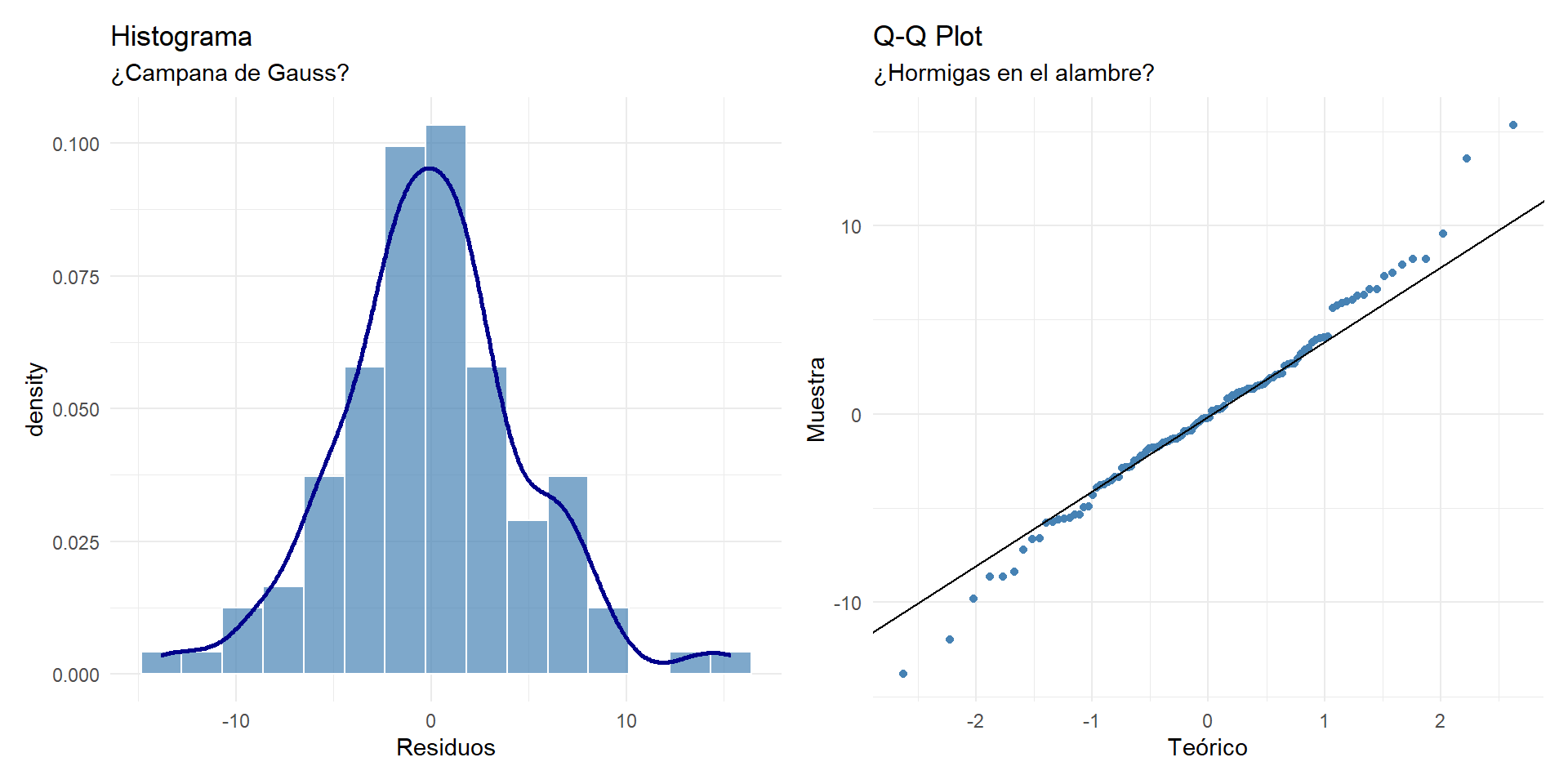
# Test formal de Shapiro-Wilk
shapiro.test(residuos)
Shapiro-Wilk normality test
data: residuos
W = 0.98644, p-value = 0.2976Con los estudentizados (vemos que práticamente iguales)…
residuos_std <- rstudent(modelo1) # Usamos residuos estudentizados
# Gráficos ggplot
p1 <- ggplot(data.frame(res = residuos_std), aes(x = res)) +
geom_histogram(aes(y = ..density..), bins = 15, fill = "steelblue", color = "white", alpha = 0.7) +
geom_density(color = "darkblue", size = 1) +
labs(title = "Histograma", subtitle = "¿Campana de Gauss?", x = "Residuos Estudentizados") + theme_minimal()
p2 <- ggplot(data.frame(res = residuos_std), aes(sample = res)) +
stat_qq(color = "steelblue") + stat_qq_line() +
labs(title = "Q-Q Plot", subtitle = "¿Hormigas en el alambre?", x = "Teórico", y = "Muestra") + theme_minimal()
p1 + p2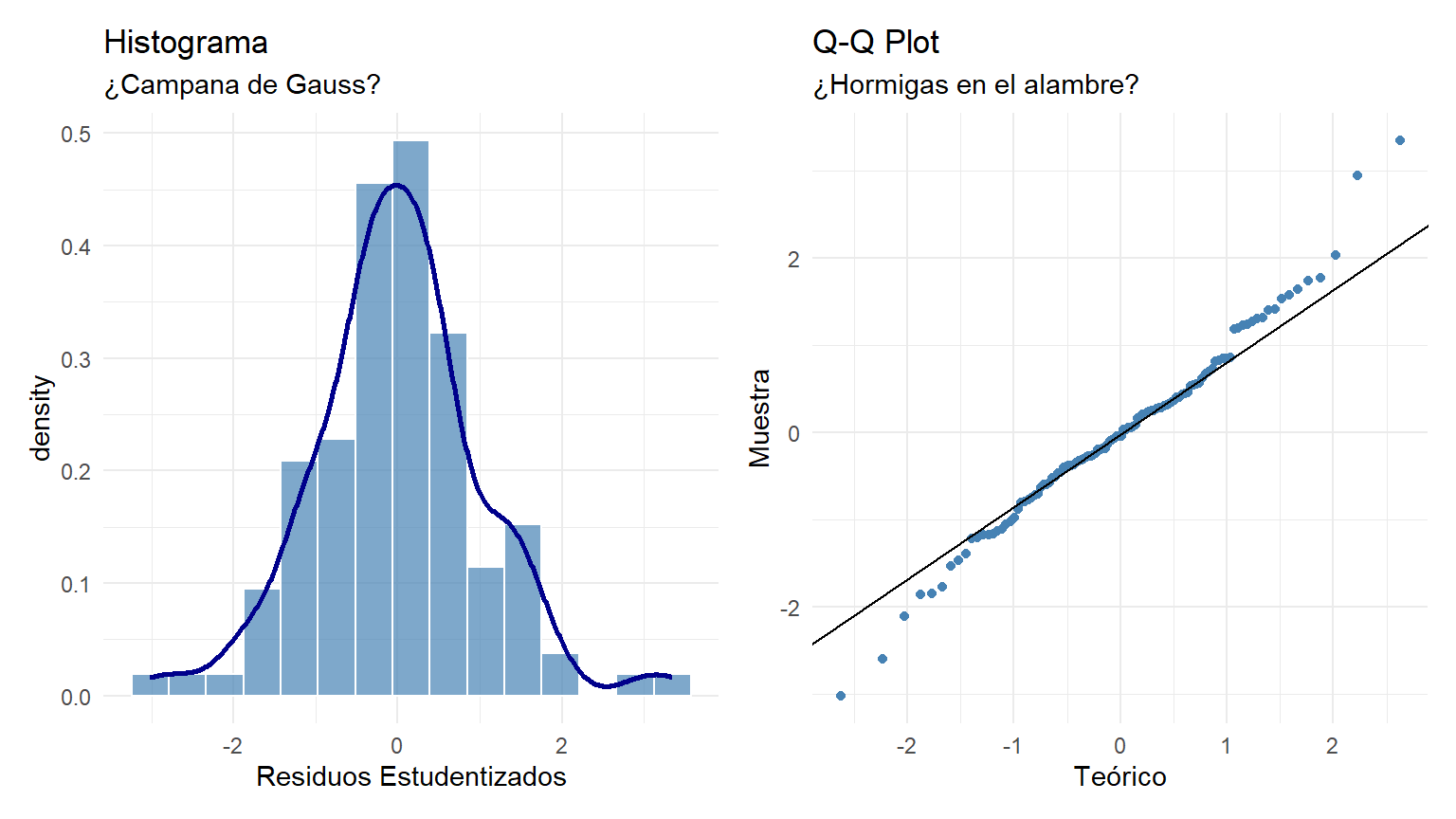
# Test formal de Shapiro-Wilk
shapiro.test(residuos_std)
Shapiro-Wilk normality test
data: residuos_std
W = 0.98432, p-value = 0.1959Diagnóstico: * Visual: El histograma es simétrico y en el Q-Q plot los puntos (“hormigas”) marchan sobre la línea diagonal sin desviaciones graves en forma de arco. * Test: El test de Shapiro-Wilk da un p-valor > 0.05, lo que confirma que los residuos son estadísticamente normales.
La precisión del modelo debe ser igual para países pobres que para ricos (varianza constante).
df_diag <- data.frame(pred = fitted(modelo1), res = residuos_std)
ggplot(df_diag, aes(x = pred, y = res)) +
geom_point(color = "steelblue", alpha = 0.6) +
geom_hline(yintercept = 0, linetype = "dashed") +
geom_smooth(method = "loess", se = FALSE, color = "red", linetype="dotted") +
labs(title = "Homocedasticidad", subtitle = "Buscamos una nube rectangular, no un megáfono",
x = "Valores Predichos", y = "Residuos") + theme_minimal()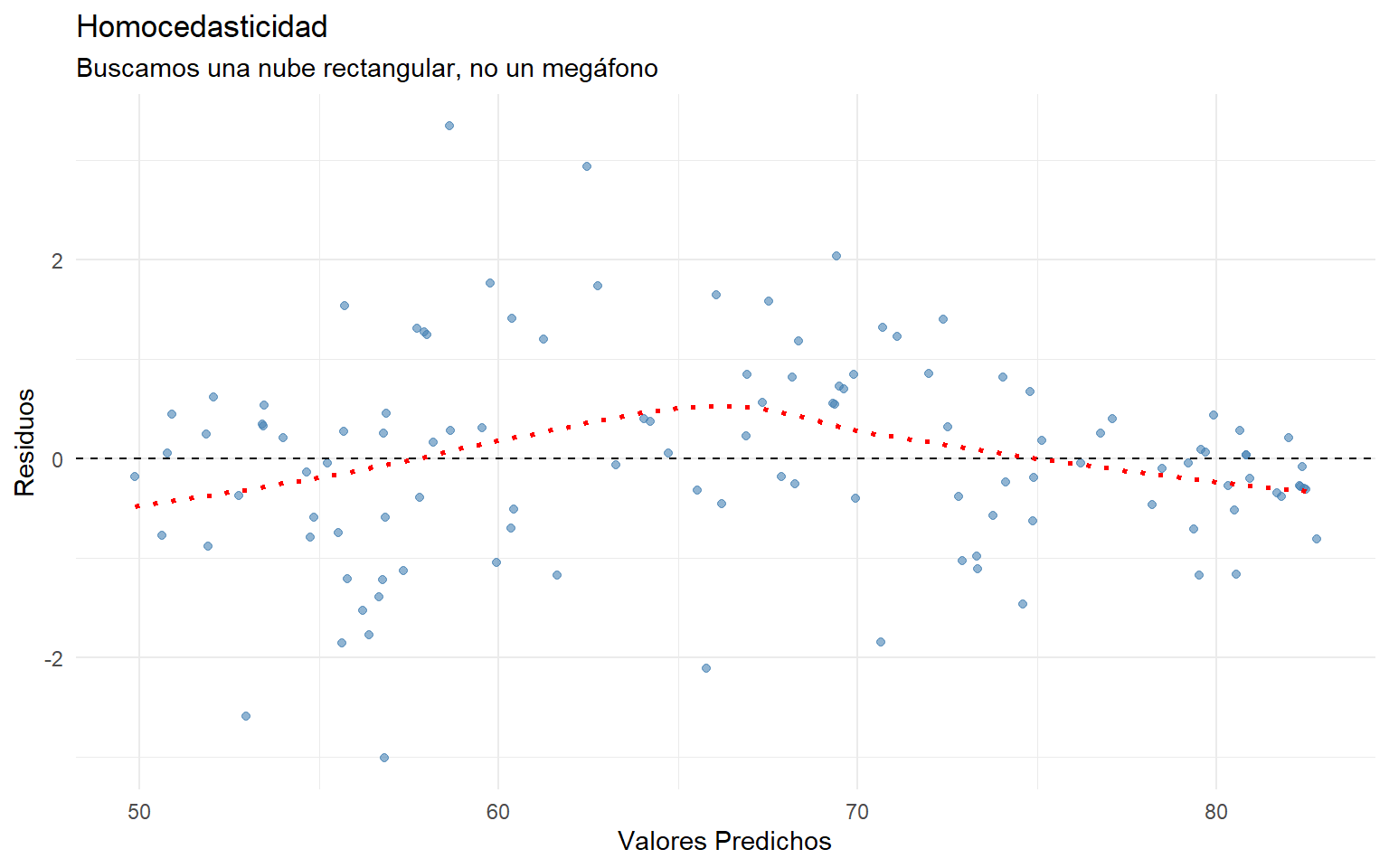
# Test de Goldfeld-Quandt
gqtest(modelo1)
Goldfeld-Quandt test
data: modelo1
GQ = 0.60476, df1 = 52, df2 = 52, p-value = 0.9637
alternative hypothesis: variance increases from segment 1 to 2Diagnóstico: * Visual: La nube es rectangular, no vemos una forma clara de “megáfono” o embudo. * Test: El test de Goldfeld-Quandt no es significativo (p > 0.05). Asumimos varianza constante.
Verificamos que no haya patrones de autocorrelación en los errores.
durbinWatsonTest(modelo1) lag Autocorrelation D-W Statistic p-value
1 0.06972319 1.858396 0.366
Alternative hypothesis: rho != 0Diagnóstico: El estadístico Durbin-Watson está muy cerca de 2 (el valor ideal). No hay problemas de independencia.
¿Están las variables independientes demasiado correlacionadas entre sí?
vif(modelo1) ln_urban ln_docs ln_hospbed ln_gdp ln_radio
2.396024 4.301509 2.568016 4.486827 2.178762 Diagnóstico Técnico: Los valores VIF son bajos (< 5), lo que indica que no hay multicolinealidad técnica grave que rompa el modelo.
Diagnóstico Lógico (Importante): Observamos en el modelo que la variable ln_hospbed (Camas) no es significativa (\(p > 0.05\)), mientras que ln_docs (Médicos) sí lo es. * ¿Significa esto que las camas de hospital no importan para salvar vidas? NO. * Si miramos la matriz de correlaciones, docs y hospbed tienen una correlación alta. * Explicación: La variable “Médicos” está capturando toda la varianza explicativa. Tener médicos implica casi siempre tener infraestructura (camas). Al introducir ambas, “Médicos” gana la batalla de la significatividad y deja a “Camas” sin nada nuevo que explicar. Es un efecto de redundancia lógica, no de falta de importancia.
Abstrayéndonos de la estadística, ¿qué nos dice este modelo sobre la realidad?
summary(modelo1)$coefficients Estimate Std. Error t value Pr(>|t|)
(Intercept) 36.8346238 4.0646893 9.0621008 5.491161e-15
ln_urban -0.3167459 1.1329809 -0.2795686 7.803335e-01
ln_docs 5.3900862 0.8219906 6.5573576 1.841574e-09
ln_hospbed 0.9306706 0.8318029 1.1188595 2.656374e-01
ln_gdp 1.7443687 0.6247760 2.7919907 6.178682e-03
ln_radio 1.6333814 0.7664704 2.1310430 3.531273e-02ln_gdp) es un predictor muy fuerte y positivo. Al ser un modelo logarítmico, indica que en países pobres, pequeños aumentos de renta disparan la esperanza de vida. En países ricos, el efecto es menor.ln_docs) es más determinante para la supervivencia femenina que la simple infraestructura física (ln_hospbed). Es el capital humano lo que salva vidas.ln_urban tiene coeficiente negativo y significativo.
ln_radio) es significativo y positivo. Esto valida la teoría de que el acceso a la información y la educación (proxy tecnológico) es un determinante social clave de la salud.Pregunta: ¿Sería posible estimar alguna clasificación de países que nos permitiera vislumbrar mejores soluciones?
Respuesta: SÍ, rotundamente.
Justificación: Hemos metido en el mismo “saco” (modelo) a países como Suiza y a países como Burundi. Aunque el modelo ajusta bien (\(R^2 \approx 0.83\)), es muy probable que la estructura de las relaciones sea diferente según el nivel de desarrollo. * En un país subdesarrollado, una cama de hospital más puede ser crítica. * En un país desarrollado, el estrés urbano o el estilo de vida pueden ser más relevantes.
Propuesta de Solución: Deberíamos realizar un Análisis Cluster (como vimos en la sesión anterior) utilizando estas mismas variables para agrupar los países en segmentos homogéneos (ej. “Desarrollados”, “Emergentes”, “En vías de desarrollo”).
Una vez segmentados, podríamos: 1. Ejecutar una regresión distinta para cada clúster. 2. Descubriríamos que los “drivers” de la esperanza de vida son diferentes en cada grupo.
Esto permitiría diseñar políticas de salud pública mucho más quirúrgicas y efectivas para cada realidad nacional.
La regresión Stepwise, aunque es muy popular en SPSS, ha sido ampliamente criticada por la comunidad estadística. Sus problemas son: 1. Alto riesgo de overfitting: El modelo se ajusta demasiado bien al conjunto de datos específico. 2. Inestabilidad: Cambios pequeños en los datos pueden llevar a modelos finales muy diferentes. 3. Resultados sesgados: Los p-valores y los intervalos de confianza del modelo final son a menudo inválidos, ya que el proceso de selección de variables es parte del mismo proceso de inferencia.
R favorece un enfoque basado en la comparación de modelos y la teoría estadística (criterios AIC/BIC) en lugar del enfoque puramente algorítmico de SPSS.
La función base de R, step(), realiza una selección de modelos basada en el Criterio de Información de Akaike (AIC), que es un método riguroso para equilibrar la bondad del ajuste con la complejidad del modelo.
Necesitas definir el modelo “más grande” (el full model) que contiene todas las variables predictoras que quieres considerar, y el modelo “más pequeño” (el null model) que solo contiene el intercepto.
# 1. Definir el modelo NULO (solo intercepto)
modelo_nulo <- lm(lifeexpf ~ 1, data = data_model)
# 2. Definir el modelo COMPLETO (todas las variables que quieres considerar)
modelo_completo <- lm(lifeexpf ~ ln_urban + ln_docs + ln_hospbed + ln_gdp + ln_radio, data = data_model) step()Ahora, usas step() para indicarle a R que pruebe los modelos, moviéndose entre el modelo nulo y el completo.
# 3. Ejecutar la selección paso a paso: 'Backward' (Hacia Atrás)
modelo_final_backward <- stats::step(modelo_completo, direction = "backward", trace = 0)
# 4. Ejecutar la selección paso a paso: 'Forward' (Hacia Adelante)
modelo_final_forward <- stats::step(modelo_nulo,
scope = list(lower = modelo_nulo, upper = modelo_completo),
direction = "forward",
trace = 0)
# 5. Ejecutar la selección paso a paso: 'Both' (Ambos sentidos)
# - Es el más común. Puede añadir o eliminar variables en cada paso.
modelo_final_both <- stats::step(modelo_nulo,
scope = list(lower = modelo_nulo, upper = modelo_completo),
direction = "both",
trace = 0)
# El resultado es el modelo final optimizado según el criterio AIC
print("--- Modelo Final Sugerido (Ambos Sentidos) ---")NA [1] "--- Modelo Final Sugerido (Ambos Sentidos) ---"summary(modelo_final_both)NA
NA Call:
NA lm(formula = lifeexpf ~ ln_docs + ln_gdp + ln_radio, data = data_model)
NA
NA Residuals:
NA female life expectancy
NA Min 1Q Median 3Q Max
NA -14.3878 -2.7412 0.1205 2.5523 16.0712
NA
NA Coefficients:
NA Estimate Std. Error t value Pr(>|t|)
NA (Intercept) 36.8682 2.8832 12.787 < 2e-16 ***
NA ln_docs 5.5669 0.7145 7.792 3.65e-12 ***
NA ln_gdp 1.8938 0.5897 3.212 0.00172 **
NA ln_radio 1.7415 0.7534 2.311 0.02264 *
NA ---
NA Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
NA
NA Residual standard error: 4.861 on 112 degrees of freedom
NA Multiple R-squared: 0.8151, Adjusted R-squared: 0.8102
NA F-statistic: 164.6 on 3 and 112 DF, p-value: < 2.2e-16print("--- Criterio de Información de Akaike ---")NA [1] "--- Criterio de Información de Akaike ---"AIC(modelo_final_both)NA [1] 701.9913print("--- Criterio de Información Bayesiano ---")NA [1] "--- Criterio de Información Bayesiano ---"BIC(modelo_final_both)NA [1] 715.7592| Característica | SPSS Stepwise | R step() |
|---|---|---|
| Criterio de Selección | p-valor (Significancia del coeficiente) | AIC (Criterio de Información de Akaike) |
| Mecanismo de Selección | Explicito (basado en p-valor y F-to-enter/remove) | Implícito (basado en la minimización del AIC) |
| Método | Forward, Backward, Stepwise |
direction = "forward", "backward", "both" |
| Función en R | N/A (se considera estadísticamente inferior) | step() (en el paquete stats de R base) |
El proceso de R (step()) es el análogo funcional del método de SPSS, pero está basado en un criterio estadístico más sólido y ampliamente aceptado (el AIC).
Para realizar una regresión paso a paso en R, usa la función step(). Esto te dará el modelo que minimiza el AIC, que es el modelo más parsimonioso (el más simple y con la mejor capacidad predictiva) que R puede encontrar.