---
title: "Inferencia paramétrica y no paramétrica"
subtitle: "Iniciando el camino"
---
## Introducciòn
Cargamos los paquetes necesarios y los datos. Usaremos el archivo `testapp.sav` proporcionado.
- `dplyr`: para manipulación de datos.
- `psych`: para análisis psicométricos y descriptivos.
- `nortest`: para tests de normalidad.
- `coin`: para tests no paramétricos.
- `expss`: para tablas de resumen y tests de significancia.
- `kableExtra`: para formateo de tablas.
- `purrr`: para consolidar en una tabla de resultados las pruebas inferenciales.
```{r setup}
#| message: false
#| warning: false
# Carga de paquetes
source('script.R')
# Opciones globales
options(scipen = 9, width = 100)
# Carga de datos desde el archivo CSV proporcionado
data <- read_spss("data/testapp.sav")
# Mostramos las primeras 5 filas
kable(head(data, 5)) %>%
kable_styling(bootstrap_options = 'responsive')
```
## Descripción de las escalas de medida
Las escalas de las variables en la base de datos son:
- **ID_CASE**: Ordinal (o nominal si el orden no importa).
- **LOADING, SPEED, SECURITY, PRIVACY, DESIGN, RESPONSIVE, MANAGEMENT, INSTSETT**: Métricas (de intervalo).
- **OS, AGENCY**: Nominales.
## Cálculo de descriptivos básicos
Existen múltiples formas de hacerlo. La más básica, sin necesidad de usar un paquete externo, es con `summary()`.
```{r summary}
summary(data)
```
Sin embargo, el paquete `psych` nos da una salida más estructurada y completa en forma de tabla, muy utilizada en ciencias sociales.
```{r describe}
describe(select(data,2:9))
```
## Determinación de la normalidad y transformaciones
Realizamos el test de Kolmogorov-Smirnov con la corrección de Lilliefors sobre todas las variables métricas. Observaremos su p-valor para determinar si podemos asumir normalidad. Del mismo modo adjuntamos la prueba Shapiro-Wilk con la misma caracterización y estructura. Recordemos que Shapiro Wilk tiene una mayor potencia y "precisión" en los resultados.
**Hipótesis:**
* H~0~: La distribución de la variable **es normal**.
* H~1~: La distribución de la variable **no es normal**.
```{r normality}
#| warning: false
#lillie.test(data$LOADING)
#lillie.test(data$SPEED)
#lillie.test(data$SECURITY)
#lillie.test(data$PRIVACY)
#lillie.test(data$DESIGN)
#lillie.test(data$RESPONSIVE)
#lillie.test(data$MANAGEMENT)
#lillie.test(data$INSTSETT)
## ---------
#shapiro.test(data$LOADING)
#shapiro.test(data$SPEED)
#shapiro.test(data$SECURITY)
#shapiro.test(data$PRIVACY)
#shapiro.test(data$DESIGN)
#shapiro.test(data$RESPONSIVE)
#shapiro.test(data$MANAGEMENT)
#shapiro.test(data$INSTSETT)
# Definir el vector de nombres de las columnas a analizar
columnas_a_testear <- c(
"LOADING",
"SPEED",
"SECURITY",
"PRIVACY",
"DESIGN",
"RESPONSIVE",
"MANAGEMENT",
"INSTSETT"
)
tabla_resultados <- map_dfr(columnas_a_testear, function(col_name) {
# Extraer el vector de datos de la columna actual
vector_datos <- data[[col_name]]
# Aplicar los tests y extraer el p-valor
# Usamos tryCatch para que si un test falla, no detenga el proceso
p_valor_lillie <- tryCatch({
lillie.test(vector_datos)$p.value
}, error = function(e) {
NA_real_
})
p_valor_shapiro <- tryCatch({
shapiro.test(vector_datos)$p.value
}, error = function(e) {
NA_real_
})
# Devolver un tibble (un dataframe moderno) con una fila de resultados
tibble(
Variable = col_name,
`P-valor KS-Lilliefors` = p_valor_lillie,
`P-valor Shapiro-Wilk` = p_valor_shapiro
)
})
# Mostrar la tabla de resultados de forma elegante
# Usamos kable y kable_styling para una presentación profesional.
tabla_resultados %>%
# Añadimos una columna para la decisión (asumiendo alpha = 0.05)
mutate(`Normalidad (Shapiro-Wilk)` = ifelse(`P-valor Shapiro-Wilk` < 0.05, "No", "Sí")) %>%
# Formateamos los números para que sean más legibles
mutate(across(where(is.numeric), ~ format.pval(., digits = 3, eps = 0.001))) %>%
kable(caption = "Resultados de los Tests de Normalidad") %>%
kable_styling(
bootstrap_options = c("striped", "hover", "condensed"),
full_width = FALSE
)
```
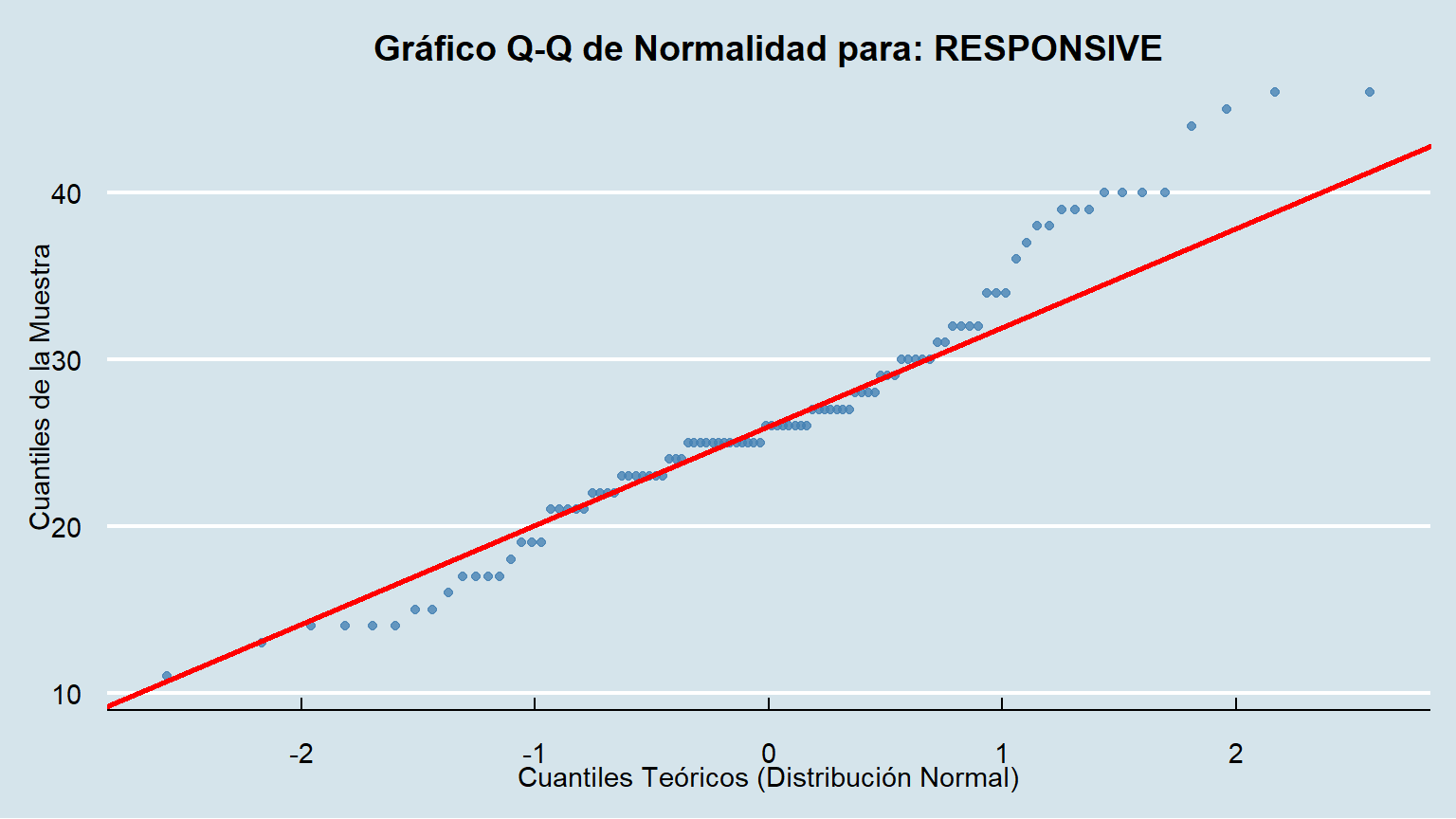
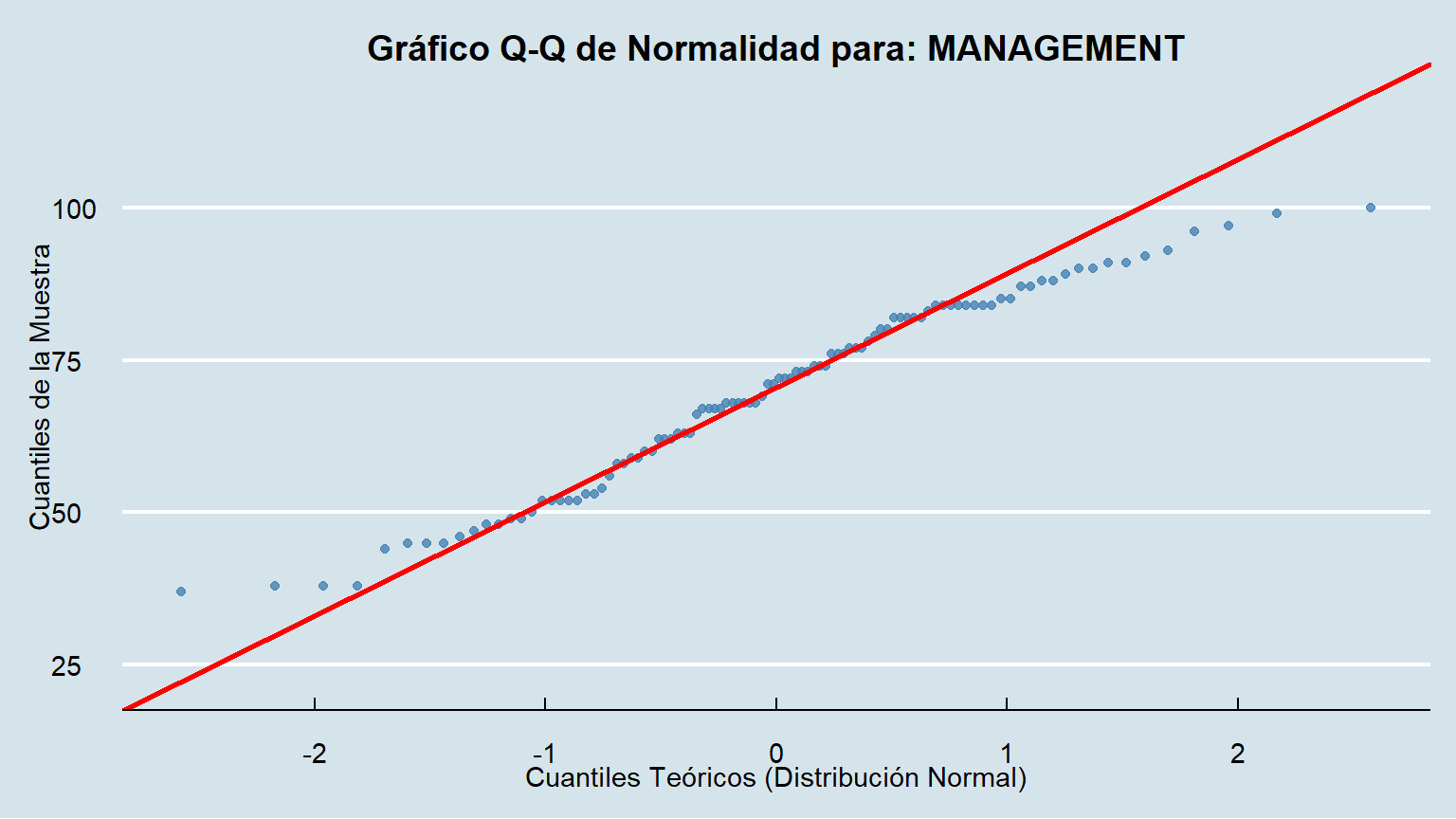
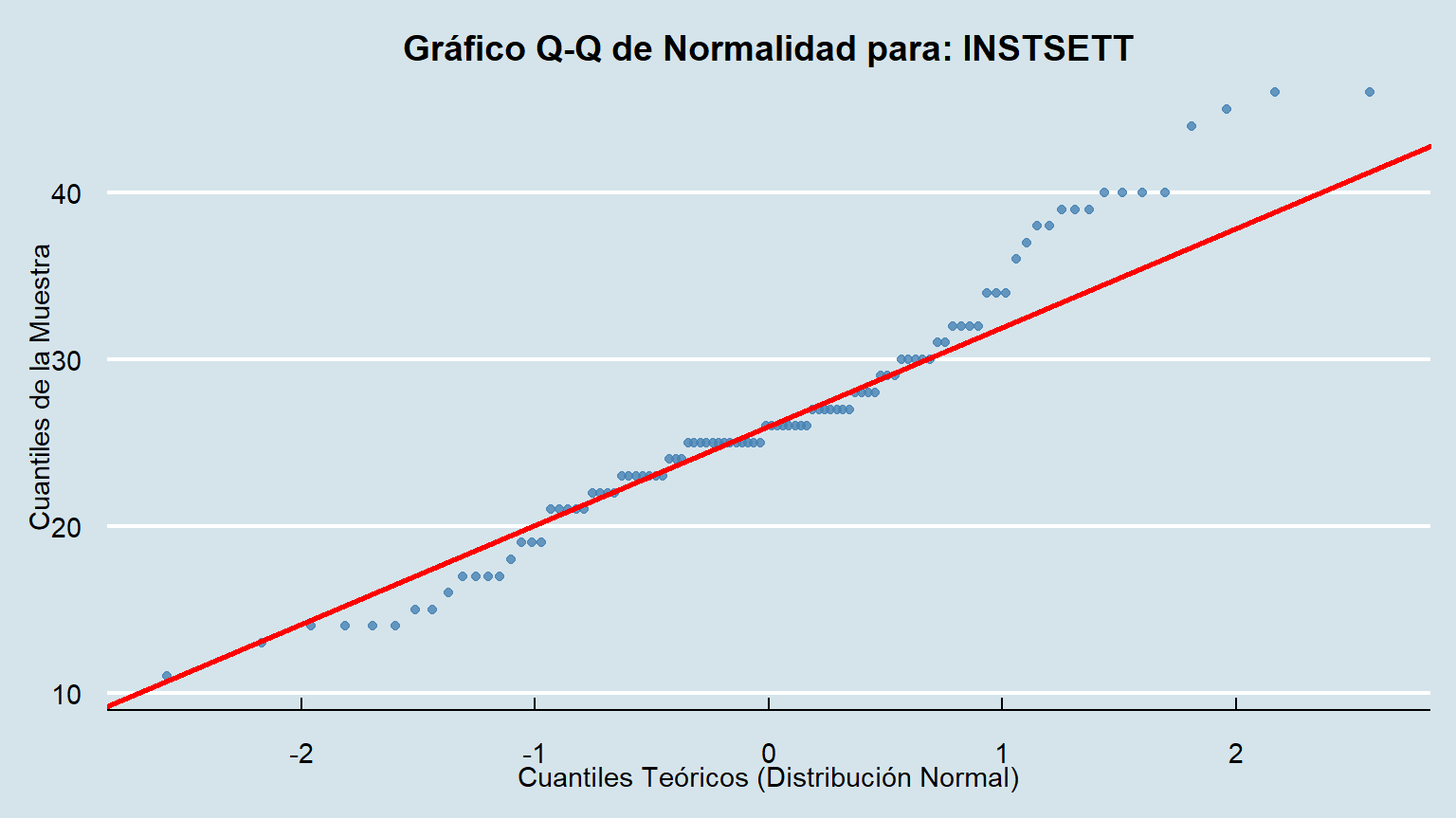
**Conclusión:** Con un nivel de significancia de 0.05, si el p-valor es menor que 0.05, rechazamos la hipótesis nula (H~0~). Vemos que para las variables `RESPONSIVE`, `MANAGEMENT` e `INSTSETT`, sus p-valores son muy bajos, por lo que no podemos considerar que sus distribuciones sean normales.
A continuación, realizamos transformaciones (raíz cuadrada, logaritmo base 10 y logaritmo neperiano) sobre estas tres variables para intentar normalizarlas.
```{r transformation}
#| warning: false
# Creación de variables transformadas
data$SQRTRESPONSIVE <- sqrt(data$RESPONSIVE)
data$SQRTMANAGEMENT <- sqrt(data$MANAGEMENT)
data$SQRTINSTSETT <- sqrt(data$INSTSETT)
data$LOGRESPONSIVE <- log(data$RESPONSIVE + 1, 10)
data$LOGMANAGEMENT <- log(data$MANAGEMENT + 1, 10)
data$LOGINSTSETT <- log(data$INSTSETT + 1, 10)
data$LNRESPONSIVE <- log(data$RESPONSIVE)
data$LNMANAGEMENT <- log(data$MANAGEMENT)
data$LNINSTSETT <- log(data$INSTSETT)
# Nuevas pruebas de normalidad
#lillie.test(data$SQRTRESPONSIVE)
#lillie.test(data$SQRTMANAGEMENT)
#lillie.test(data$SQRTINSTSETT)
#lillie.test(data$LOGRESPONSIVE)
#lillie.test(data$LOGMANAGEMENT)
#lillie.test(data$LOGINSTSETT)
#lillie.test(data$LNRESPONSIVE)
#lillie.test(data$LNMANAGEMENT)
#lillie.test(data$LNINSTSETT)
## ---
#shapiro.test(data$SQRTRESPONSIVE)
#shapiro.test(data$SQRTMANAGEMENT)
#shapiro.test(data$SQRTINSTSETT)
#shapiro.test(data$LOGRESPONSIVE)
#shapiro.test(data$LOGMANAGEMENT)
#shapiro.test(data$LOGINSTSETT)
#shapiro.test(data$LNRESPONSIVE)
#shapiro.test(data$LNMANAGEMENT)
#shapiro.test(data$LNINSTSETT)
# 1. Definir el vector de nombres de las columnas a analizar
columnas_a_testear <- c(
"SQRTRESPONSIVE",
"SQRTMANAGEMENT",
"SQRTINSTSETT",
"LOGRESPONSIVE",
"LOGMANAGEMENT",
"LOGINSTSETT",
"LNRESPONSIVE",
"LNMANAGEMENT",
"LNINSTSETT"
)
library(purrr)
tabla_resultados <- map_dfr(columnas_a_testear, function(col_name) {
# Extraer el vector de datos de la columna actual
vector_datos <- data[[col_name]]
# Aplicar los tests y extraer el p-valor
# Usamos tryCatch para que si un test falla, no detenga el proceso
p_valor_lillie <- tryCatch({
lillie.test(vector_datos)$p.value
}, error = function(e) {
NA_real_
})
p_valor_shapiro <- tryCatch({
shapiro.test(vector_datos)$p.value
}, error = function(e) {
NA_real_
})
# Devolver un tibble (un dataframe moderno) con una fila de resultados
tibble(
Variable = col_name,
`P-valor KS-Lilliefors` = p_valor_lillie,
`P-valor Shapiro-Wilk` = p_valor_shapiro
)
})
# 3. Mostrar la tabla de resultados de forma elegante
# Usamos kable y kable_styling para una presentación profesional.
tabla_resultados %>%
# Añadimos una columna para la decisión (asumiendo alpha = 0.05)
mutate(`Normalidad (Shapiro-Wilk)` = ifelse(`P-valor Shapiro-Wilk` < 0.05, "No", "Sí")) %>%
# Formateamos los números para que sean más legibles
mutate(across(where(is.numeric), ~ format.pval(., digits = 3, eps = 0.001))) %>%
kable(caption = "Resultados de los Tests de Normalidad") %>%
kable_styling(
bootstrap_options = c("striped", "hover", "condensed"),
full_width = FALSE
)
```
**Conclusión:** Tras realizar las transformaciones, observamos que los p-valores de los tests de normalidad siguen siendo significativamente bajos. Aunque alguno de ellos nos permitiría no rechazar la normalidad, creeemos más acertado dado que existe divergencia entre las dos pruebas, ser muy conservadores en este aspecto. Por lo tanto, ninguna de las transformaciones lineales ha conseguido normalizar estas variables. Deberemos tratarlas como no paramétricas.
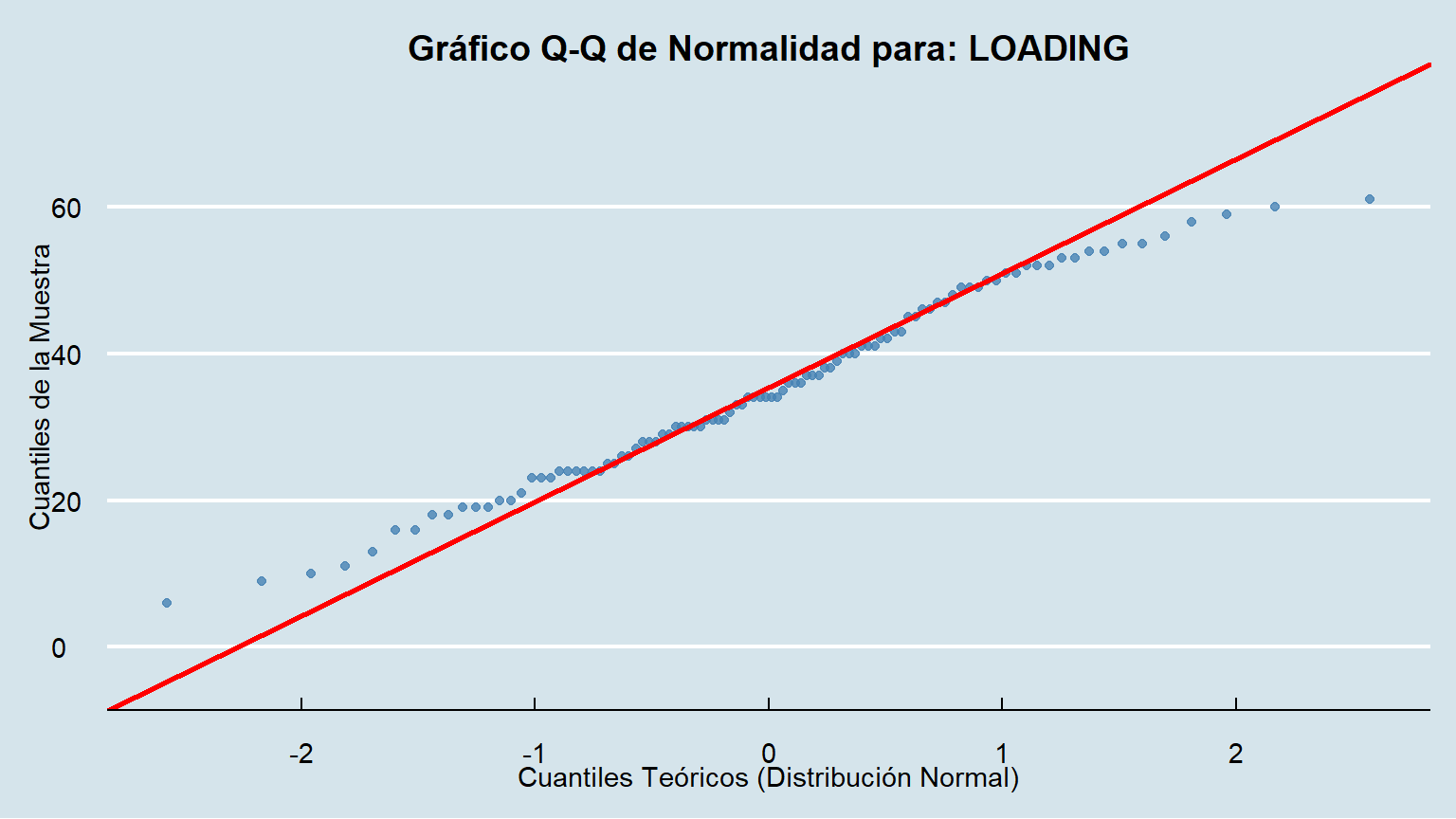
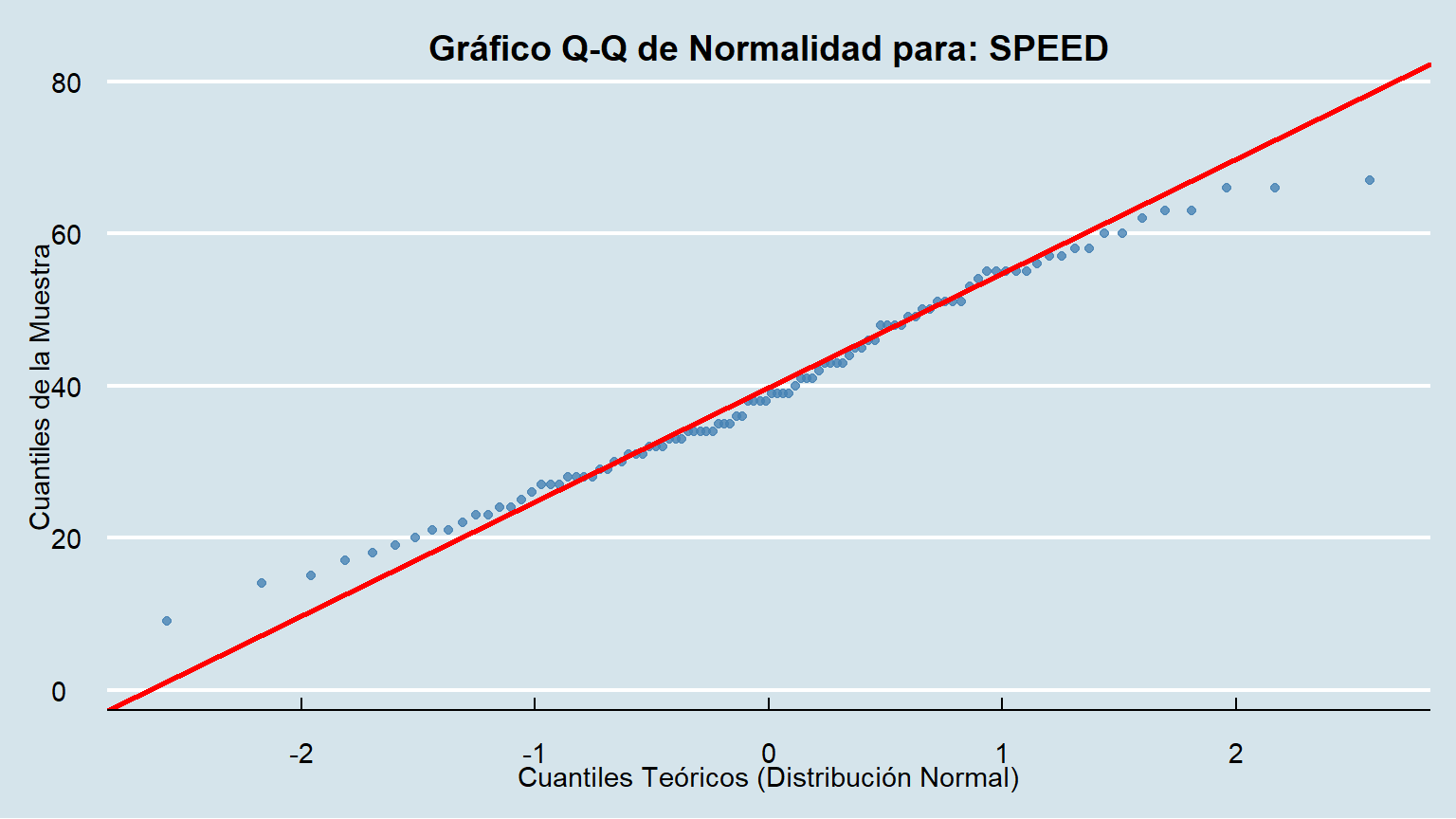
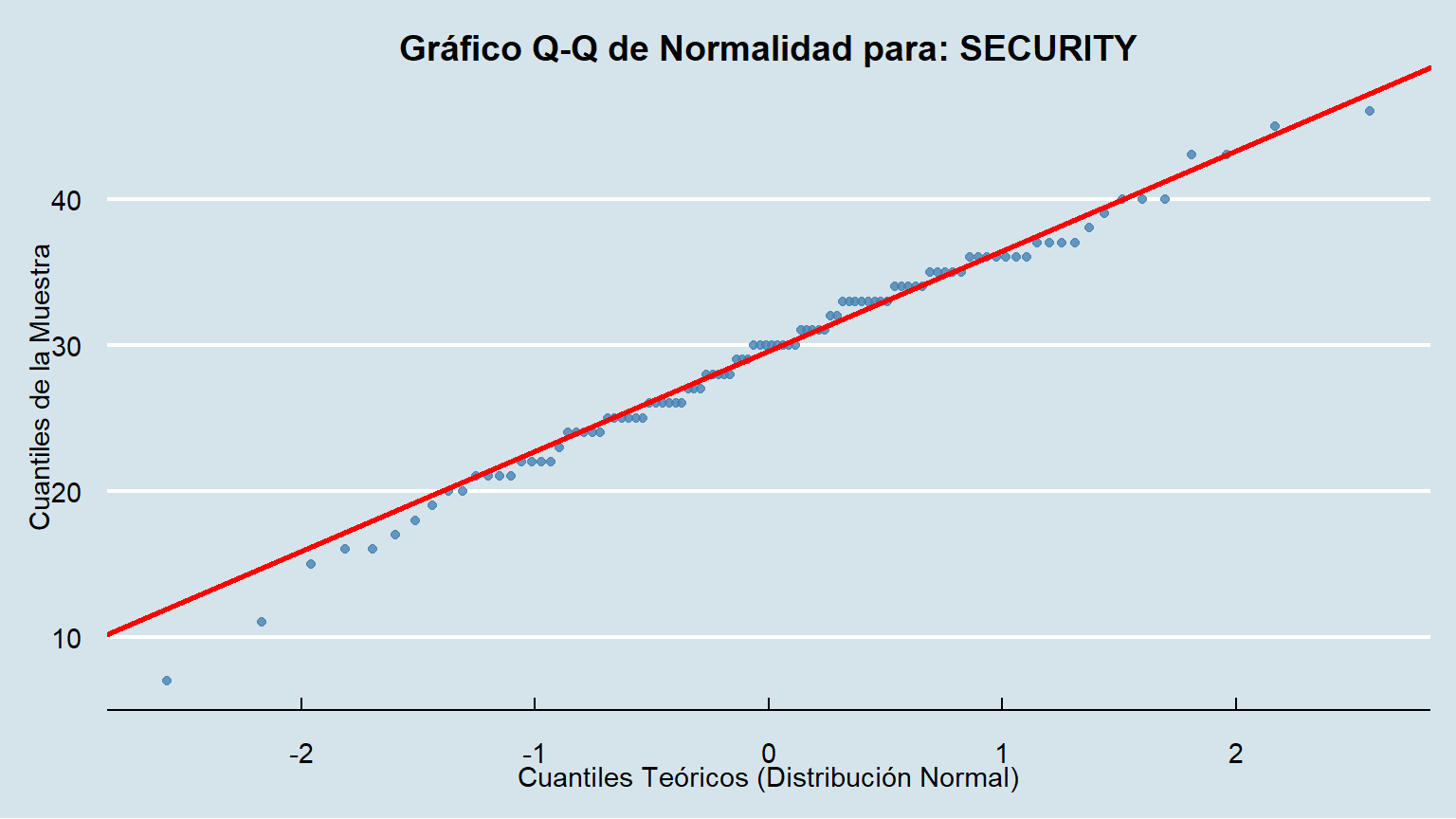
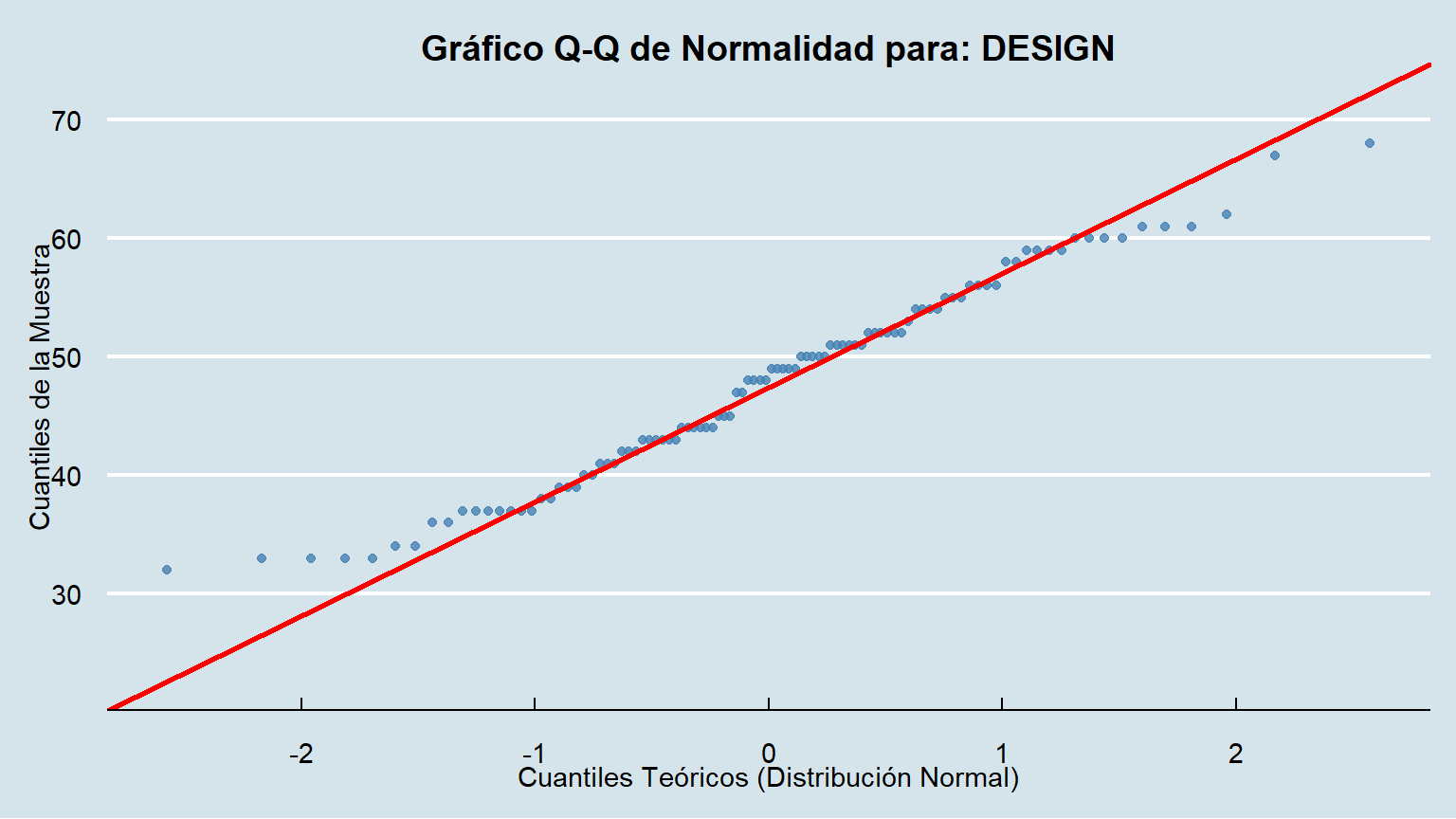
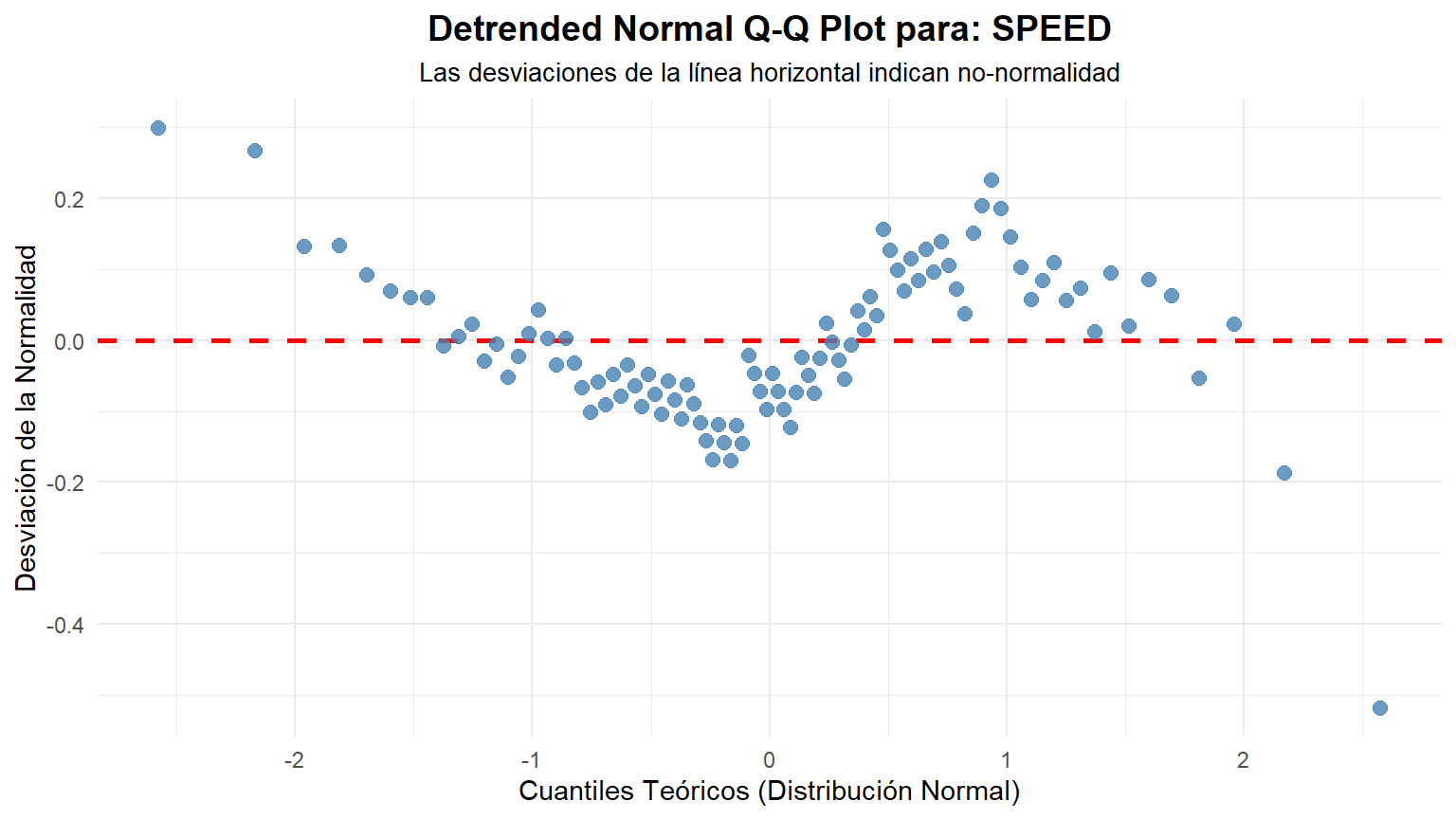
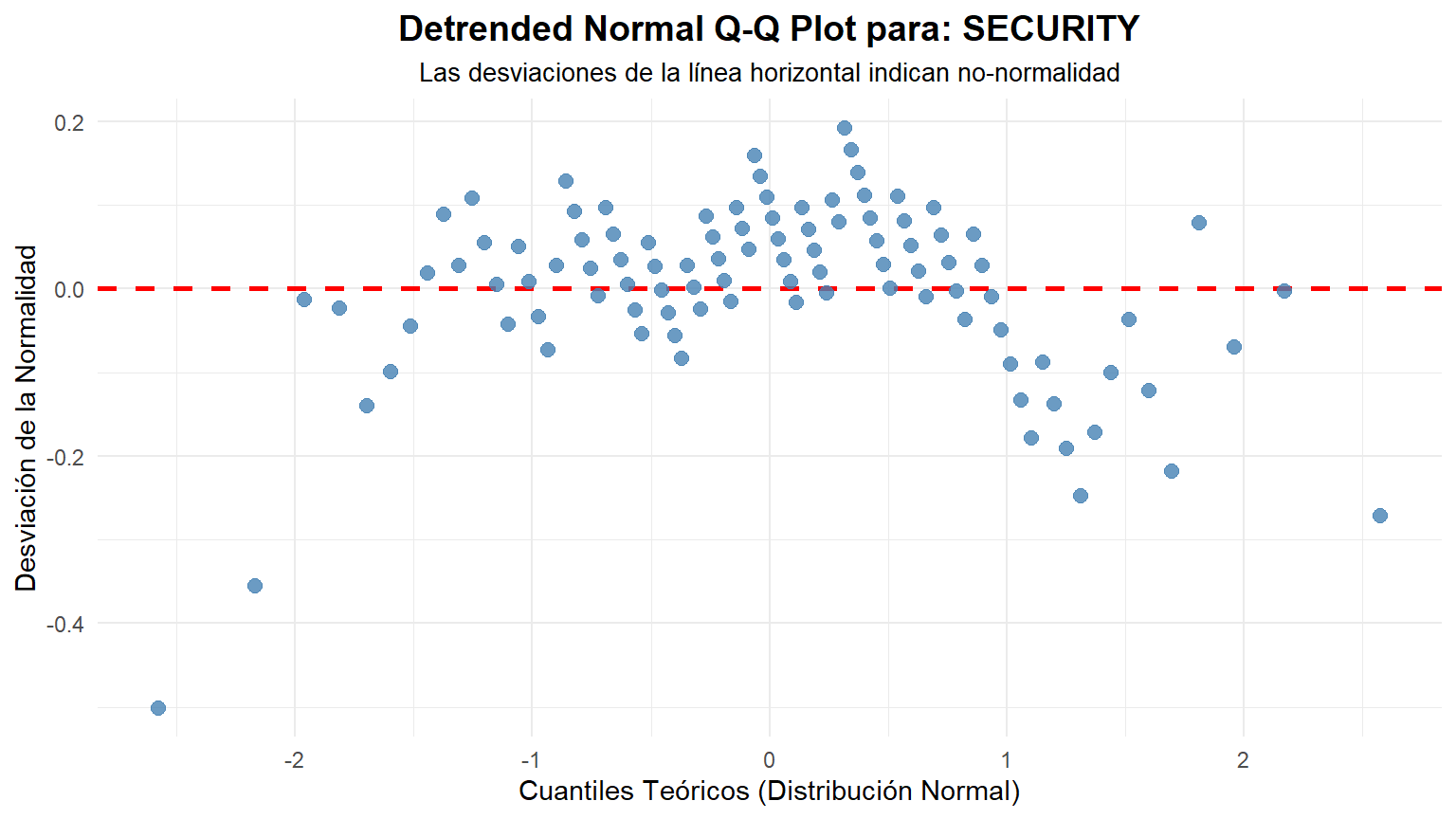
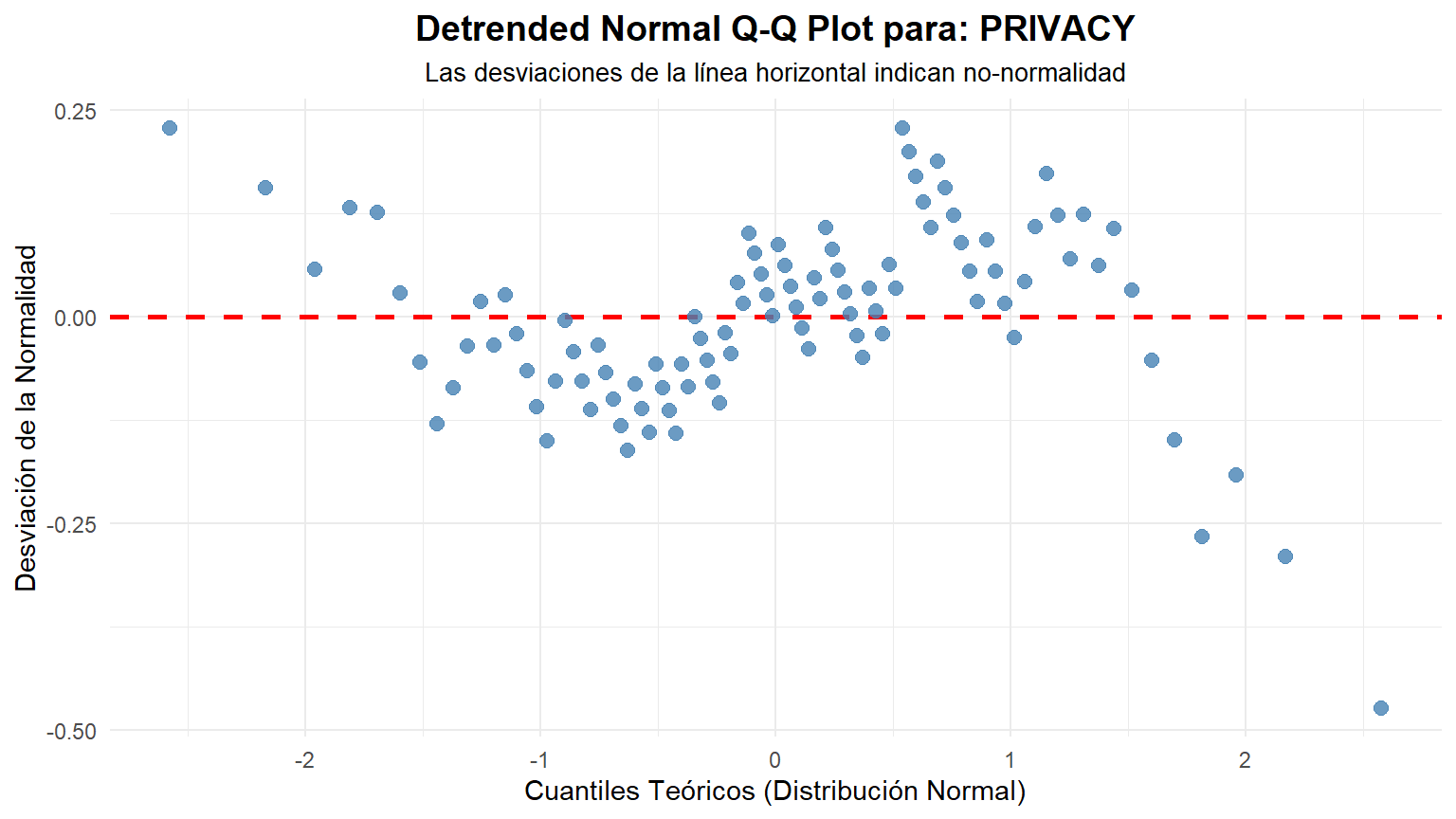
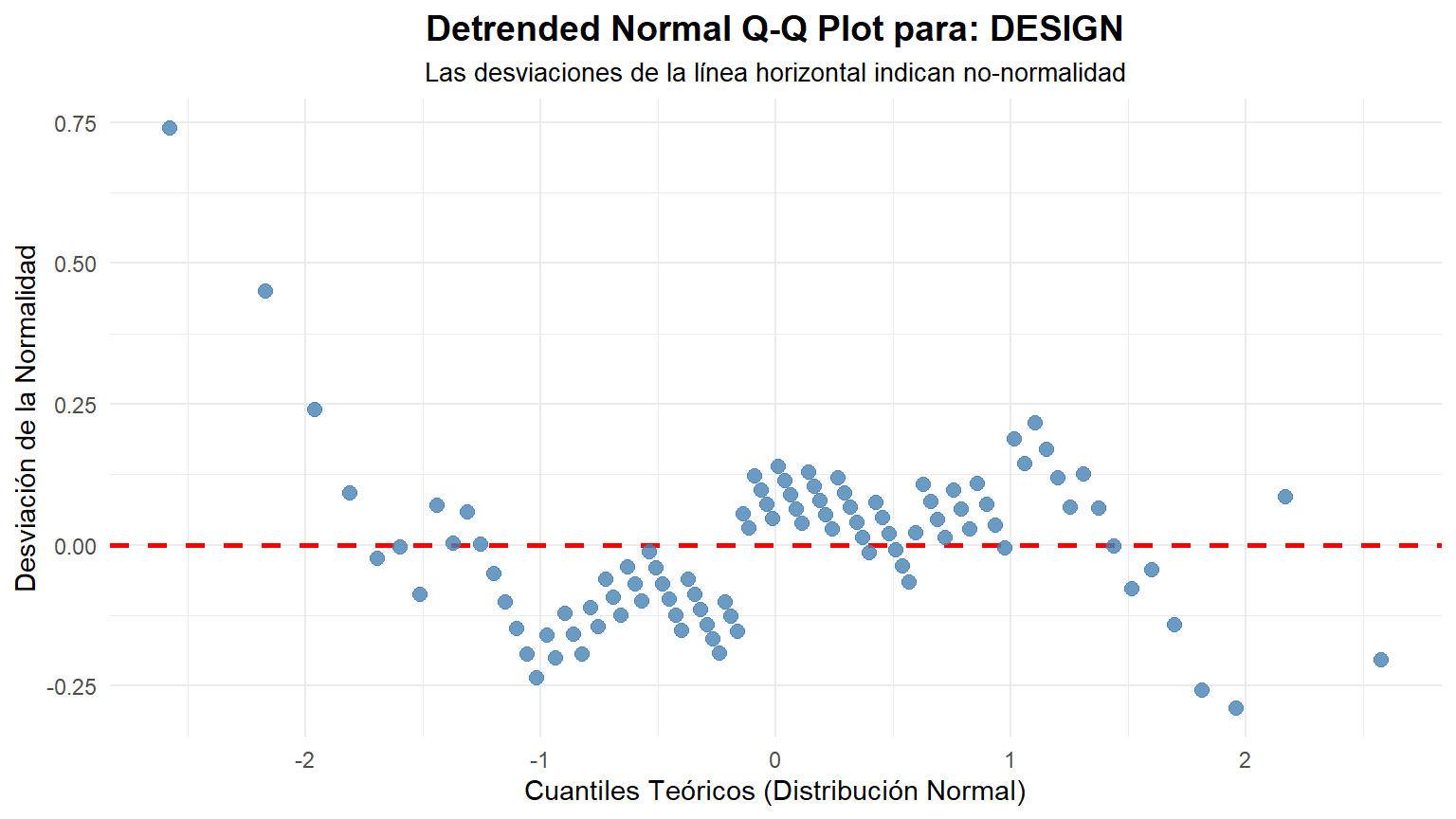
Para acompañar el análisis, cremos los gráficos QQPLOT ...
```{r qqplot}
#| fig-height: 4.5
#| fig-width: 8
#' Crea un gráfico Q-Q de normalidad para una columna específica de un dataframe.
#'
#' @param data El dataframe que contiene los datos.
#' @param column El nombre de la columna a analizar. Se puede pasar sin comillas
#' (ej. mi_columna) o como una cadena de texto (ej. "mi_columna").
#'
#' @return Un objeto de ggplot que representa el gráfico Q-Q.
#'
create_qqplot <- function(data, column) {
col_name_str <- rlang::as_name(rlang::enquo(column))
# Comprobamos que la columna exista en el dataframe
if (!col_name_str %in% names(data)) {
stop(paste("La columna '", col_name_str, "' no se encuentra en el dataframe."))
}
# --- Creación del Gráfico ---
p <- ggplot(data, aes(sample = {{ column }})) +
# El operador {{ }} es la forma moderna de pasar un argumento de función a aes()
geom_qq(color = "steelblue", alpha = 0.8) + # Dibuja los puntos
geom_qq_line(color = "red", linewidth = 1) + # Dibuja la línea de referencia
# Personalización de títulos y etiquetas
labs(
title = paste("Gráfico Q-Q de Normalidad para:", col_name_str),
x = "Cuantiles Teóricos (Distribución Normal)",
y = "Cuantiles de la Muestra"
) +
# Tema limpio
ggthemes::theme_economist() +
theme(
plot.title = element_text(hjust = 0.5, face = "bold", size = 14)
)
return(p)
}
create_qqplot(data = data, column = LOADING)
create_qqplot(data = data, column = SPEED)
create_qqplot(data = data, column = SECURITY)
create_qqplot(data = data, column = PRIVACY)
create_qqplot(data = data, column = DESIGN)
create_qqplot(data = data, column = RESPONSIVE)
create_qqplot(data = data, column = MANAGEMENT)
create_qqplot(data = data, column = INSTSETT)
```
### Interpretación Visual
- Si los puntos azules se ajustan estrechamente a la línea roja, es una fuerte indicación de que los datos provienen de una distribución normal.
- Si los puntos se desvían sistemáticamente de la línea (formando una curva), los datos no son normales.
```{r qqplot_detrended}
#| fig-height: 4.5
#| fig-width: 8
#' Crea un gráfico "Detrended Normal Q-Q" para una columna específica.
#'
#' Este gráfico muestra las desviaciones de la normalidad, replicando el
#' "Detrended Normal Q-Q Plot" de SPSS.
#'
#' @param data El dataframe que contiene los datos.
#' @param column El nombre de la columna a analizar (sin comillas).
#'
#' @return Un objeto de ggplot que representa el gráfico.
#'
create_detrended_qqplot <- function(data, column) {
# --- 1. Capturar y validar la entrada ---
col_name_str <- rlang::as_name(rlang::enquo(column))
if (!col_name_str %in% names(data)) {
stop(paste("La columna '", col_name_str, "' no se encuentra en el dataframe."))
}
# Extraer el vector de datos y eliminar los NAs
y <- data[[col_name_str]]
y <- y[!is.na(y)]
# --- 2. Calcular los componentes del Q-Q plot ---
# Ordenar los datos de la muestra
y_sorted <- sort(y)
# Calcular los cuantiles teóricos de una distribución normal estándar
n <- length(y)
theoretical_quantiles <- qnorm(ppoints(n))
# Estandarizar los datos de la muestra para que sean comparables
# (media 0, desviación estándar 1)
y_standardized <- (y_sorted - mean(y)) / sd(y)
# --- 3. ¡LA MAGIA! Calcular la "detrended" difference ---
# La diferencia es simplemente el cuantil de la muestra menos el cuantil teórico
detrended_difference <- y_standardized - theoretical_quantiles
# Crear un dataframe para ggplot
plot_data <- data.frame(
theoretical = theoretical_quantiles,
difference = detrended_difference
)
# --- 4. Creación del Gráfico ---
p <- ggplot(plot_data, aes(x = theoretical, y = difference)) +
# Añadimos una línea de referencia horizontal en y=0
geom_hline(yintercept = 0, color = "red", linewidth = 1, linetype = "dashed") +
# Dibujamos los puntos de la diferencia
geom_point(color = "steelblue", alpha = 0.8, size = 2.5) +
# Personalización de títulos y etiquetas
labs(
title = paste("Detrended Normal Q-Q Plot para:", col_name_str),
subtitle = "Las desviaciones de la línea horizontal indican no-normalidad",
x = "Cuantiles Teóricos (Distribución Normal)",
y = "Desviación de la Normalidad"
) +
# Tema limpio
theme_minimal() +
theme(
plot.title = element_text(hjust = 0.5, face = "bold", size = 14),
plot.subtitle = element_text(hjust = 0.5, size = 10)
)
return(p)
}
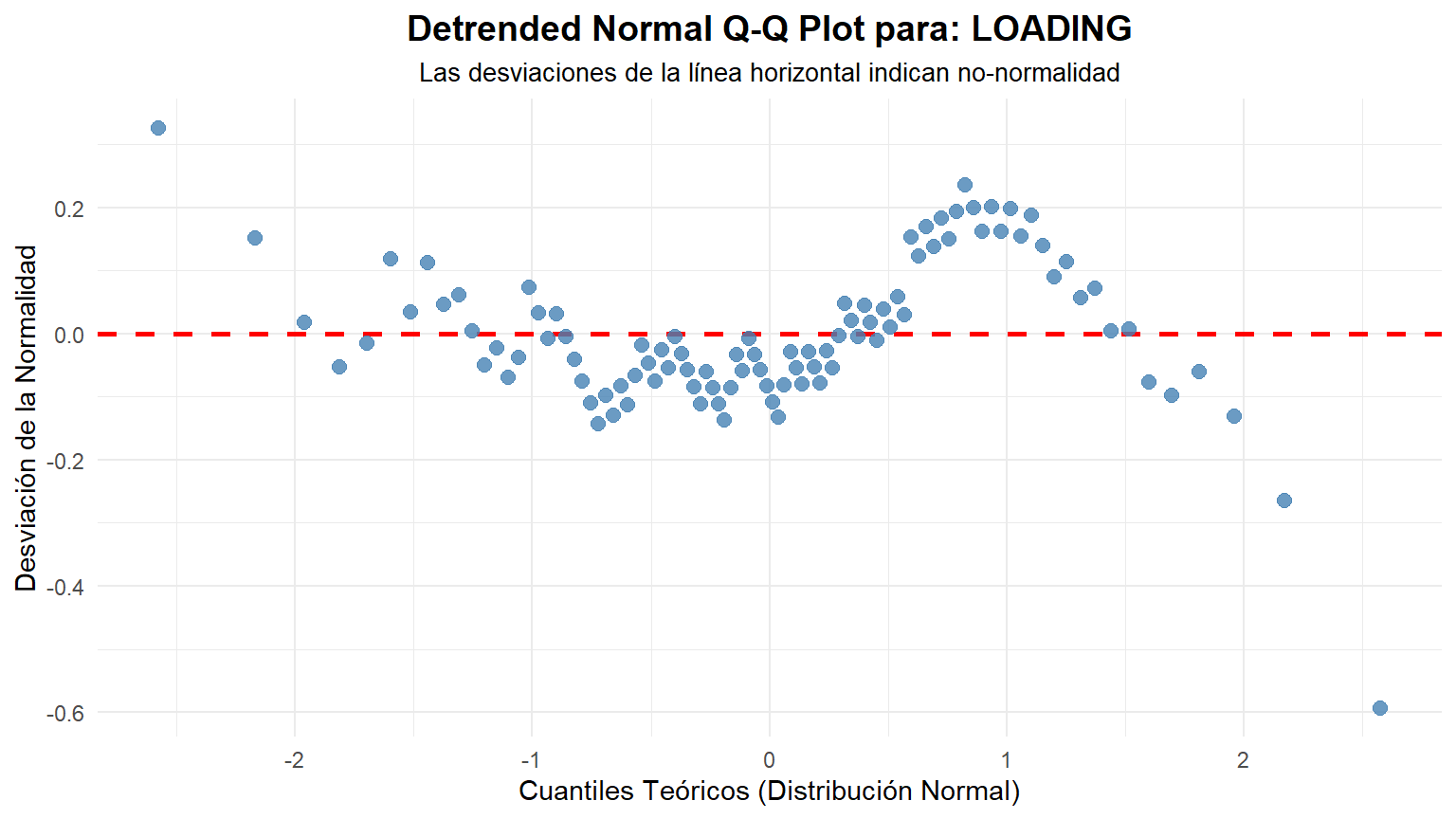
create_detrended_qqplot(data = data, column = LOADING)
create_detrended_qqplot(data = data, column = SPEED)
create_detrended_qqplot(data = data, column = SECURITY)
create_detrended_qqplot(data = data, column = PRIVACY)
create_detrended_qqplot(data = data, column = DESIGN)
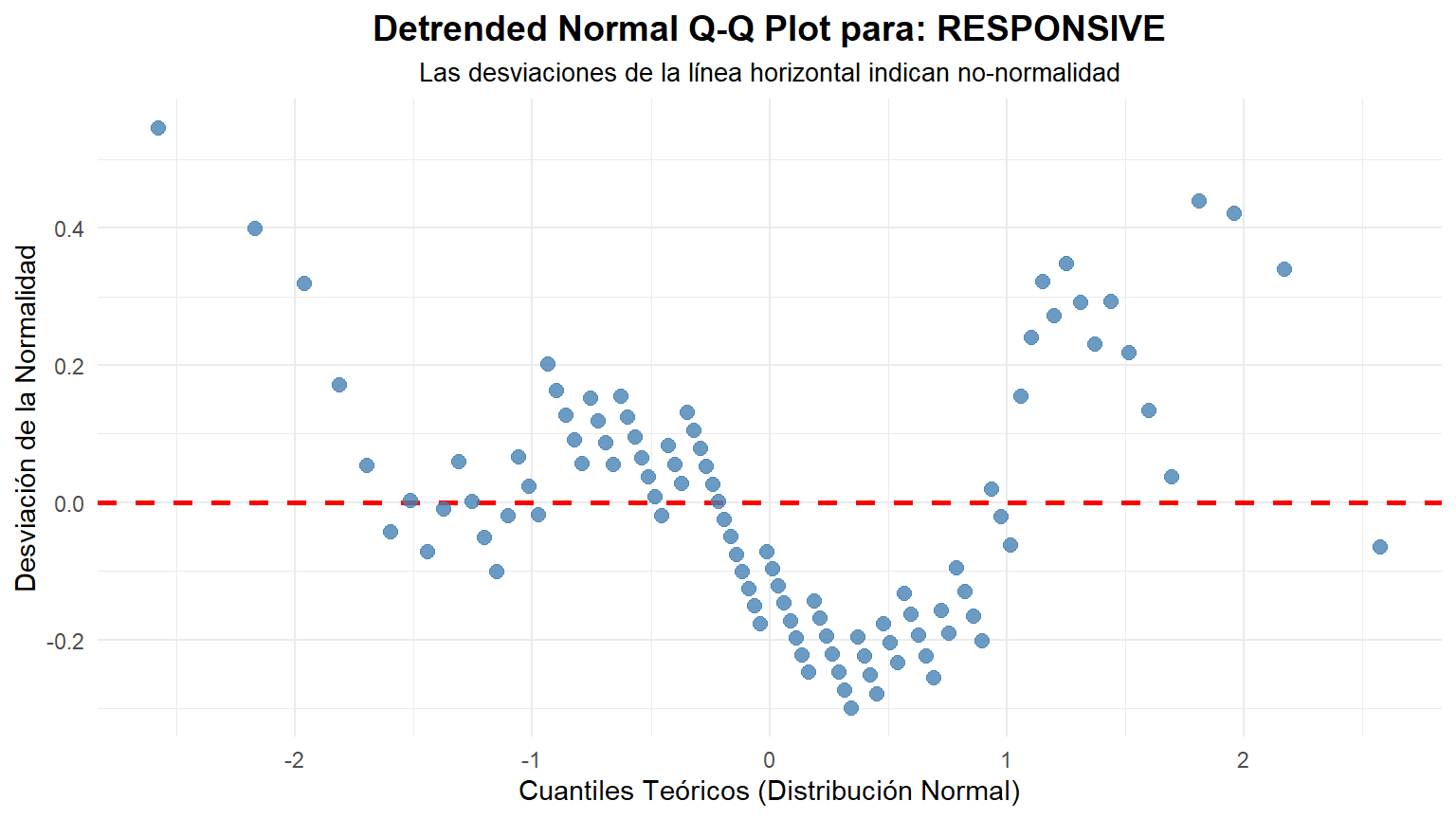
create_detrended_qqplot(data = data, column = RESPONSIVE)
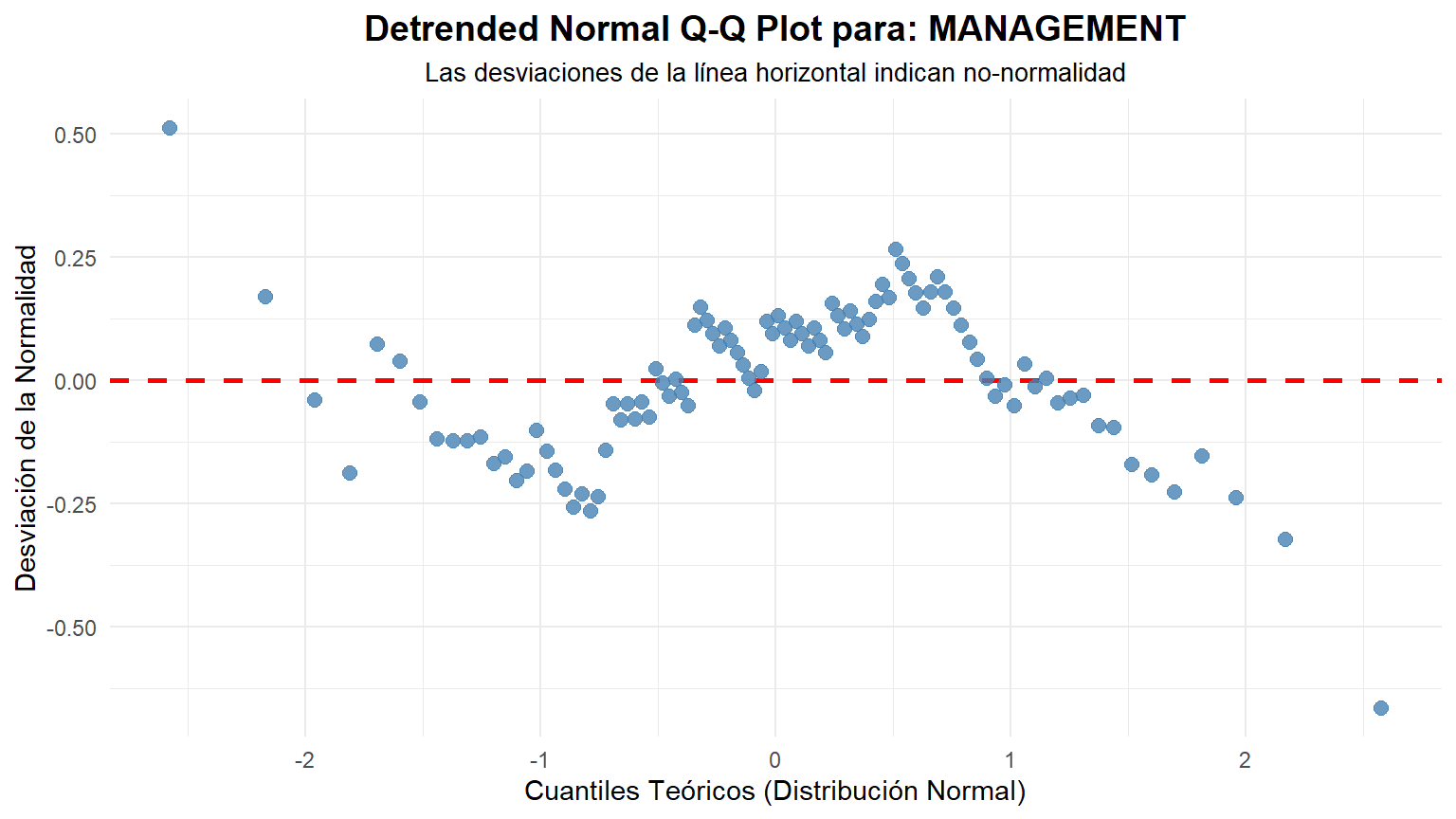
create_detrended_qqplot(data = data, column = MANAGEMENT)
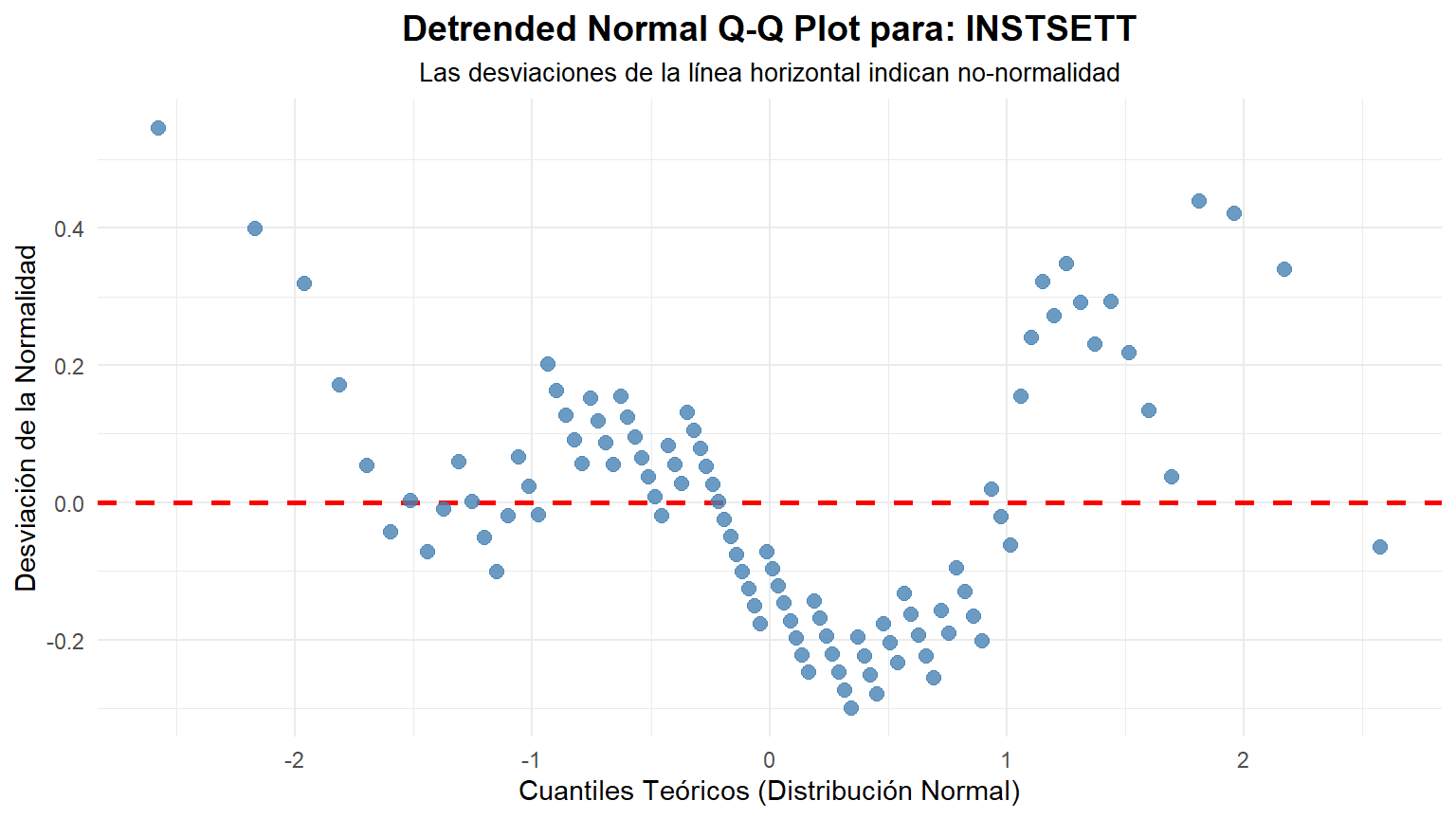
create_detrended_qqplot(data = data, column = INSTSETT)
```
### Interpretación Visual del Gráfico "Detrended"
- Si los datos son normales: Los puntos se distribuirán de forma aleatoria y sin un patrón claro alrededor de la línea horizontal roja en y=0. No deberían formar ninguna curva o estructura discernible.
- Si los datos NO son normales: Los puntos formarán un patrón sistemático. Por ejemplo:
- Asimetría a la derecha (Right Skew): Los puntos formarán una curva en forma de "U" o de sonrisa.
- Asimetría a la izquierda (Left Skew): Los puntos formarán una curva en forma de "U" invertida o de ceño fruncido.
- Colas pesadas (Heavy Tails): Los puntos formarán una "S" invertida.
- Colas ligeras (Light Tails): Los puntos formarán una "S" normal.
## Diferencias de medias por Sistema Operativo (OS)
Para determinar si existen diferencias en las medias de las variables métricas con respecto a los grupos de `OS`, usaremos el paquete `expss` para generar una tabla de medias y realizar un test de significancia (ANOVA en este caso).
```{r}
#| message: false
#| warning: false
data %>%
tab_cols(total(), OS) %>%
tab_cells(LOADING, SPEED, SECURITY, PRIVACY, DESIGN) %>%
tab_stat_mean_sd_n() %>%
tab_last_sig_means(keep = 'means') %>%
tab_pivot() %>%
as.datatable_widget()
```
**Conclusión Comercial:** Se observan diferencias estadísticamente significativas en las valoraciones de `LOADING`, `SPEED`, `PRIVACY` y `DESIGN` entre los diferentes sistemas operativos. La variable `SECURITY` es la única que no presenta diferencias significativas. Analizando las medias, podemos ver qué sistema operativo obtiene mejores o peores valoraciones en cada aspecto, lo que puede guiar esfuerzos de optimización específicos para cada plataforma.
También podríamos haberlo hecho siguiendo las pautas naturales de Levene test y posteriormente t-test para todos los pares (SPSS). Esto se debería repetir para todos los pares de OS y para todas las variables.
```{r}
#| message: false
#| warning: false
test_levene <- leveneTest(LOADING ~ factor(OS), data = filter(data, OS %in% c(1,2)))
print(test_levene)
p_valor_levene <- test_levene$`Pr(>F)`[1]
if (p_valor_levene >= 0.05) {
print("Resultado de Levene (p >= 0.05): Las varianzas se asumen iguales.\n")
print("Ejecutando t-test de Student (var.equal = TRUE).\n\n")
test_t <- t.test(LOADING ~ factor(OS), data = filter(data, OS %in% c(1,2)), var.equal = TRUE)
} else {
print("Resultado de Levene (p < 0.05): Las varianzas se asumen diferentes.\n")
print("Ejecutando t-test de Welch (var.equal = FALSE).\n\n")
test_t <- t.test(LOADING ~ factor(OS), data = filter(data, OS %in% c(1,2)), var.equal = FALSE)
}
print(test_t)
```
## Diferencias en variables no normales por Tipo de Tester (AGENCY)
Dado que `RESPONSIVE`, `MANAGEMENT` e `INSTSETT` no siguen una distribución normal, para comparar las diferencias entre los dos grupos de `AGENCY` debemos usar un test no paramétrico. El test adecuado es el **Test de Wilcoxon-Mann-Whitney** (equivalente al `t.test` para datos no paramétricos).
```{r}
wilcox.test(data$RESPONSIVE ~ data$AGENCY, data = data, distribution = "exact", conf.int = 0.95)
wilcox.test(data$MANAGEMENT ~ data$AGENCY, data = data, distribution = "exact", conf.int = 0.95)
wilcox.test(data$INSTSETT ~ data$AGENCY, data = data, distribution = "exact", conf.int = 0.95)
```
**Conclusión Comercial:** En los tres casos, los p-valores son mayores que 0.05. Por lo tanto, no podemos rechazar la hipótesis nula de igualdad. Esto significa que **no existen diferencias estadísticamente significativas** en las valoraciones de `RESPONSIVE`, `MANAGEMENT` e `INSTSETT` entre las agencias cualitativas y cuantitativas. El tipo de agencia no parece influir en la percepción de estos atributos de la aplicación.
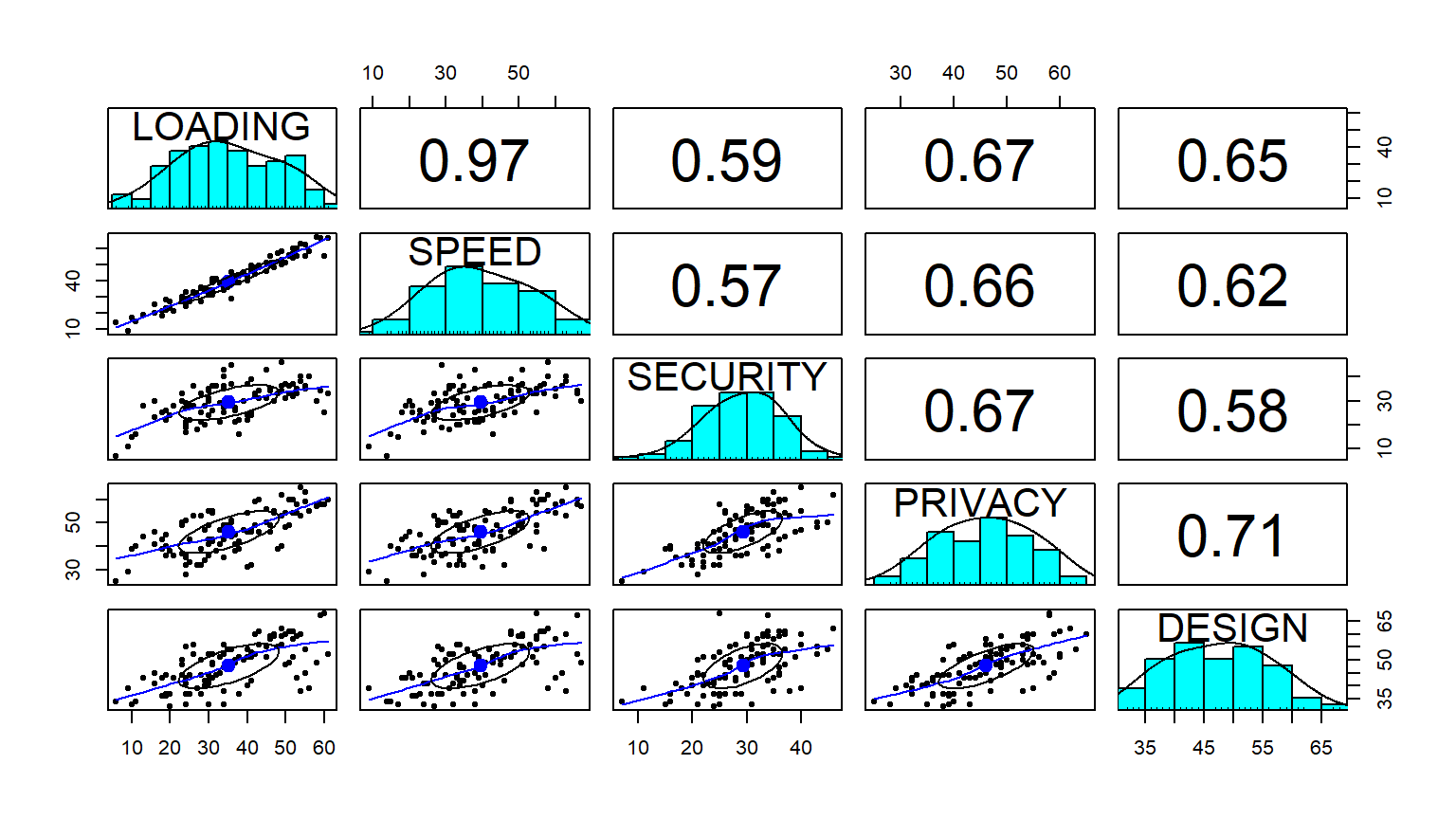
## Relación entre variables métricas con normalidad
Para analizar la relación entre las variables métricas que sí asumimos como normales (`LOADING` a `DESIGN`), calculamos el **coeficiente de correlación de Pearson**.
```{r}
#| fig-height: 4.5
#| fig-width: 8
options(scipen = 9, digits = 5, width = 9999)
# Test de correlación
corr.test(
select(data, LOADING:DESIGN),
use = "pairwise",
method = "pearson",
alpha = .01,
ci = TRUE
)
# Matriz de gráficos de dispersión y correlaciones
pairs.panels(select(data, LOADING:DESIGN))
```
## Relación entre variables métricas sin normalidad
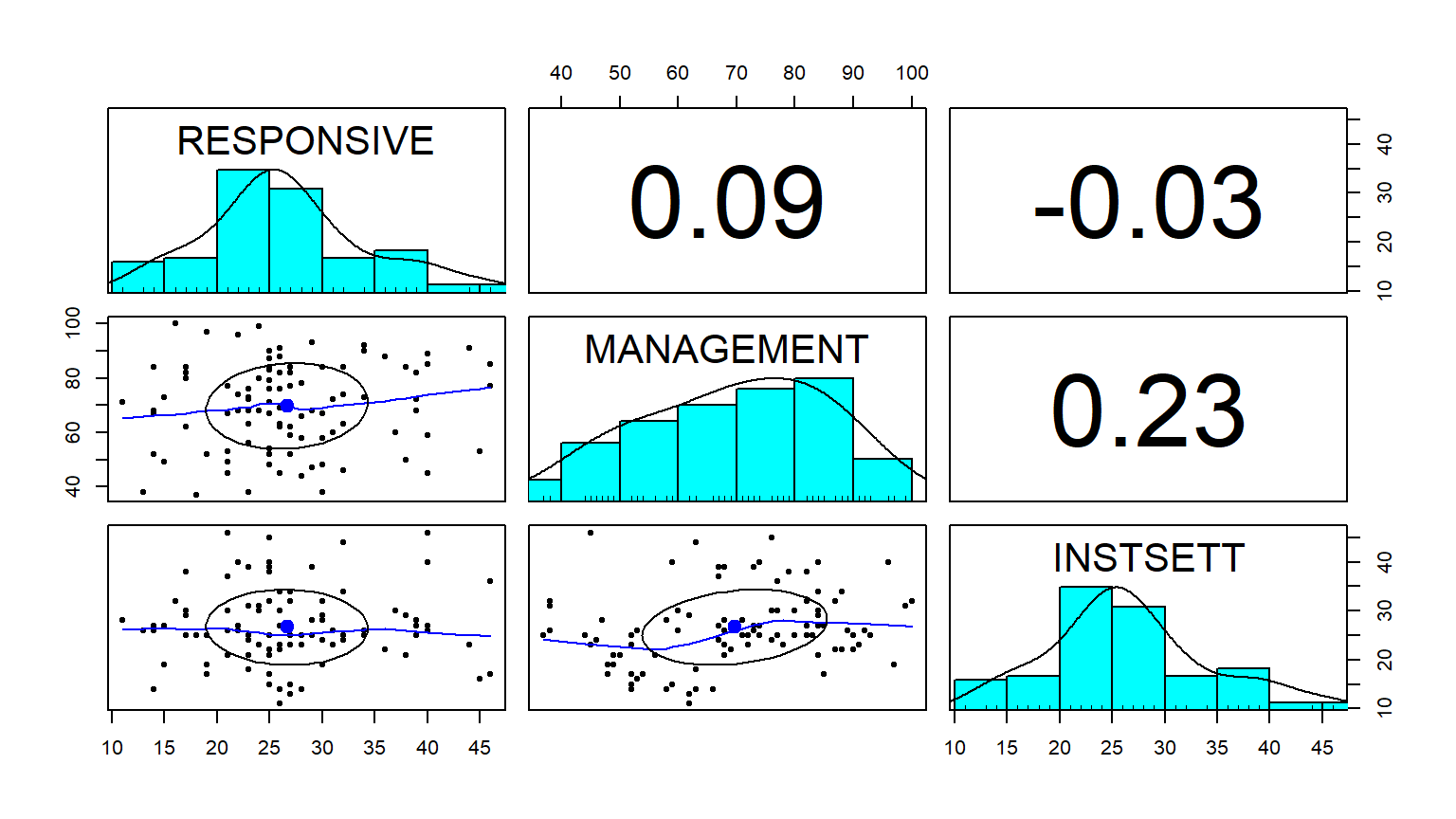
Para las variables que no son normales (`RESPONSIVE`, `MANAGEMENT`, `INSTSETT`), debemos usar coeficientes de correlación no paramétricos, como **Spearman** o **Kendall**.
```{r}
#| fig-height: 4.5
#| fig-width: 8
options(scipen = 9, digits = 5, width = 9999)
# Correlación de Spearman
corr.test(select(data, RESPONSIVE:INSTSETT), use = "pairwise", method = "spearman", alpha = .01, ci = TRUE)
pairs.panels(select(data, RESPONSIVE:INSTSETT), method = "spearman")
# Correlación de Kendall
corr.test(select(data, RESPONSIVE:INSTSETT), use = "pairwise", method = "kendall", alpha = .01, ci = TRUE)
pairs.panels(select(data, RESPONSIVE:INSTSETT), method = "kendall")
```
## Dependencia entre variables métricas recodificadas y tipo de tester
Para analizar la dependencia entre las variables no paramétricas y el tipo de tester, primero las recodificamos en tres niveles (Bajo, Medio, Alto) basándonos en sus cuartiles.
```{r}
#| message: false
#| warning: false
# Recodificación de variables
data = data %>% compute(
RRESPONSIVE = expss::recode(
RESPONSIVE,
lo %thru% fivenum(RESPONSIVE)[2] ~ 1,
(fivenum(RESPONSIVE)[2] + 0.001) %thru% fivenum(RESPONSIVE)[3] ~ 2,
(fivenum(RESPONSIVE)[3] + 0.001) %thru% hi ~ 3
),
RMANAGEMENT = expss::recode(
MANAGEMENT,
lo %thru% fivenum(MANAGEMENT)[2] ~ 1,
(fivenum(MANAGEMENT)[2] + 0.001) %thru% fivenum(MANAGEMENT)[3] ~ 2,
(fivenum(MANAGEMENT)[3] + 0.001) %thru% hi ~ 3
),
RINSTSETT = expss::recode(
INSTSETT,
lo %thru% fivenum(INSTSETT)[2] ~ 1,
(fivenum(INSTSETT)[2] + 0.001) %thru% fivenum(INSTSETT)[3] ~ 2,
(fivenum(INSTSETT)[3] + 0.001) %thru% hi ~ 3
)
)
# Aplicar etiquetas a los nuevos niveles
val_lab(data$RRESPONSIVE) = num_lab("
1 Bajo
2 Medio
3 Alto
")
val_lab(data$RMANAGEMENT) =num_lab("
1 Bajo
2 Medio
3 Alto
")
val_lab(data$RINSTSETT) = num_lab("
1 Bajo
2 Medio
3 Alto
")
# Tabla de contingencia con porcentajes y test de significancia (Chi-cuadrado)
options(digits = 3, width = 9999)
data %>%
tab_cols(total(), AGENCY) %>%
tab_cells(RRESPONSIVE, RMANAGEMENT, RINSTSETT) %>%
tab_stat_cases() %>%
tab_last_sig_cases() %>%
tab_pivot() %>%
as.datatable_widget()
```
**Conclusión:** El test de Chi-cuadrado nos indica si existe una relación de dependencia entre el nivel de valoración (Bajo, Medio, Alto) y el tipo de agencia. Si la significancia es menor a 0.05, podemos concluir que el tipo de agencia está relacionado con la probabilidad de que la valoración de ese atributo sea alta, media o baja.
Otra forma de hacerlo más estándar y utilizando R-base sería ...
```{r}
table(data$RMANAGEMENT, data$AGENCY)
chisq.test(table(data$RRESPONSIVE, data$AGENCY))
table(data$RMANAGEMENT, data$AGENCY)
chisq.test(table(data$RMANAGEMENT, data$AGENCY),correct=)
table(data$RINSTSETT, data$AGENCY)
chisq.test(table(data$RINSTSETT, data$AGENCY))
```
@R-tidyverse
@R-purrr
@R-rstatix
@R-reactable
@R-highcharter
@R-outliers
@R-EnvStats
@R-dunn.test
@R-psych
@R-FactoMineR
@R-factoextra
@R-ca
@R-gapminder
@R-corrplot
@R-patchwork
@R-performance
@R-titanic
@R-tidymodels
@R-pROC
@R-caret
@R-see
@R-psych
@R-nortest
@R-coin
@R-kableExtra
@R-car
@R-readr
@R-expss
@R-maditr
@R-DT",
@R-NbClust",
@R-cluster",
@R-expss",
@R-maditr",
@R-car",
@R-lmtest",
@R-nortest",
@R-Hmisc",
@R-patchwork"